基因芯片分型软件(AxAS)再升级,提高罕见杂合位点分型准确性!
2023-02-06 10:41:25, FBS 刘瑢 赛默飞世尔科技生命科学产品
Axiom Analysis Suite(AxAS)基因芯片分型软件至今已推出了5年, 软件从只能分析Genotyping,升级到能够同时分析CNV。今年6月份AxAS进行了第10次升级(最新版本为AxAS v5.1),本次升级主要针对罕见杂合位点分型结果的准确性进行优化。

罕见杂合位点基因分型结果出现假阳性,是使用基因芯片进行基因分型的研究者非常希望解决的问题,以往针对于罕见位点往往需要专门的生信人员进行仔细核查。为了让研究者能够更轻松的使用AxAS分析软件,经过我们研发团队不断的尝试及努力,最新版本的AxAS v5.1软件增加了罕见杂合位点分型矫正功能,此功能可以在一定程度上提高罕见杂合位点的检测准确性。具体更新内容如下:
罕见杂合位点分型结果矫正
作为本次更新最重要的一个内容,矫正的功能被整合到Best Practices Workflow和 Genotyping Workflow中,软件会自动将怀疑为假阳性的位点调整为No Call,在人类芯片中此矫正是默认的。
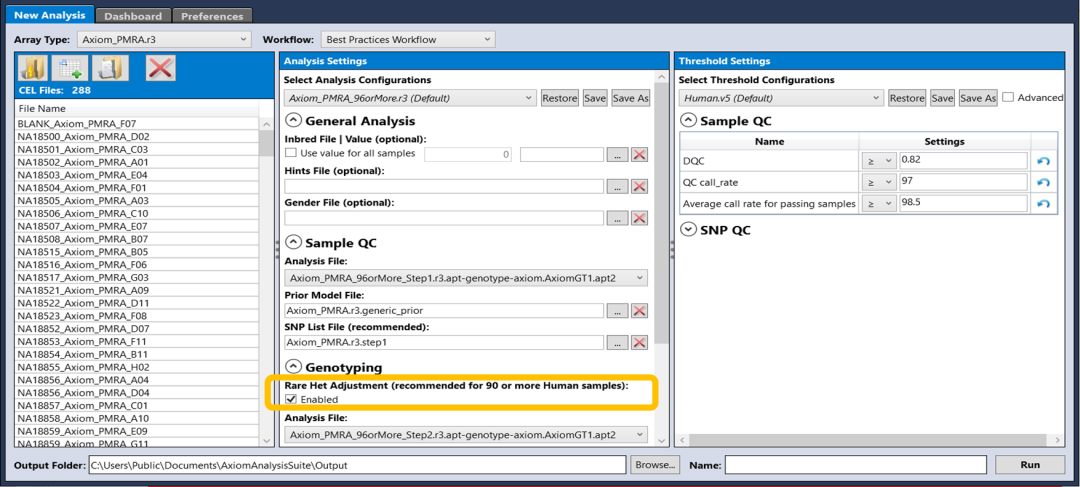
图1 罕见杂合位点调整选项
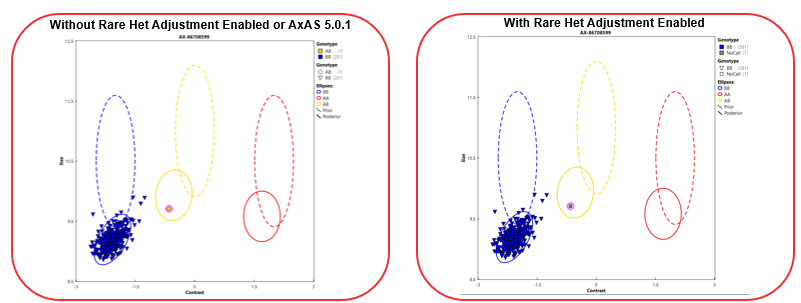
图2 罕见杂合位点经过矫正后变成No Call
矫正后的Output文件夹中会新增两个文件(AxiomGT1.rare_het.report 和 AxiomGT1.rare_het_maj_hom.report)。文件包含每个被检测位点的检测结果及参数信息。

图3 新增的output文件
注意:此功能不适用于动植物样本和少于90个人类样本的分析。
其他更新及修复
AxAS v5.1在进行多等位基因分析时为确保使用正确的先验模型文件,AxAS会进行内部检查(避免多等位基因用成二等位基因的文件) 。
AxAS v5.1对应的APT版本是2.11.3。
AxAS v5.1修复了VCF文件中InDel探针基因组位置。v5.0和v5.0.1中有些以VCF格式输出基因型的InDel 探针有可能会出现1个核苷酸的位置输出错误。
对于支持Allele Translation的芯片(Pharmacoscan, PMD Array, and derivative arrays run with Axiom 2.0 Plus Assay),即使样本中出现很多单倍型No Call也会完成translation。
用户可根据需要进行样本间一致性比较,或检测样本和参考样本位点的一致性比较。一致性分析在Sample Table QC Analysis的下拉菜单中。CEL样本名中包含()符号也可以,例如:MySample(PMDA)A1.cel。
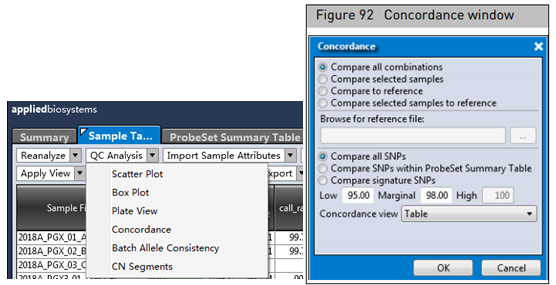
图4 使用AxAS进行一致性分析
注意事项
多等位探针导出:要导出部分SNP的基因型数据,如果没有选择注释文件,会出现多等位探针的calls丢失或改变。
解决方法:
1. 导出所有探针。
2. 导出genotypes时选择合适的注释文件。
重新分析之前使用v5.0分析过的文件,更改阈值参数可能会出现错误。
重新生成SNP Metrics时,如Posterior file选项中选择了其他的芯片的Posterior文件,软件不会报错。
PharmacoScan的分析文件包中,当改变了探针的注释文件,重新产成SNP Metrics时,AxAS Viewer可能会卡死。
通过下面的链接即可进入AxAS v5.1下载界面!
https://www.thermofisher.com/cn/zh/home/life-science/microarray-analysis/microarray-analysis-instruments-software-services/microarray-analysis-software/axiom-analysis-suite.html06-13 沃特世
时代印记,可靠之选 | Alliance HPLC经典二十八载06-13 沃特世
沃特世发布全新代谢组学和脂质组学软件06-13 沃特世
【快讯】探索无界,加速转化 | 2024“科学空间”系列前沿对话峰会之“探索与转化的加速可及”在沪成功举办06-13
后节手术: 利用术中OCT技术的好处06-13 徕卡显微系统
徕卡精准空间生物学解决方案 第二弹06-13 童昕
快速、可靠的清洁度分析解决方案06-13 徕卡显微系统
以完整的深度揭开生命的奥秘06-13 徕卡显微系统
药包材新公示首个LCMSMS法,揭秘金属涂层中“隐形杀手”双酚A06-13 分析中心
特色应用(二) | SPME-GC-MS/MS研究不同储藏年份玉米风味物质差异06-13 创新中心
岛津连续八年入选“健康经营优良法人 ~White500~”06-13 企划部
共创美好记忆 --记2024年岛津上海分公司家庭日活动06-13 岛津上海工会
岛津招聘 | 风险合规专员06-13 人事部
质谱成像大讲堂丨如何获得一张完美的组织切片?06-13 分析中心
labtech2024 正式启幕丨4大主题洞察实验室用户需求,你的议题你做主!06-13 labtech
仪器行业新机遇:全国推动大规模设备更新!06-13 Sielin
TESCAN TENSOR 4D-STEM 客户案例研讨会06-13 TESCAN中国
有奖 | 标乐面向全球征集微观结构美图06-13 标乐中国
连续流快速原子荧光技术的开创者06-13
蚊子为什么专咬我!"吗喽" 的命也是命......06-13 小 M