一文读懂PCA分析(原理、算法、可视化)内附Python作图代码
2023-06-27 21:30:41, 质谱创新组学 上海欧易生物医学科技有限公司
01
什么是PCA
PCA(Principal Component Analysis,主成分分析)是一种常用的降维技术,用于从高维数据中提取最重要的特征。它通过线性变换将原始数据映射到一个新的坐标系中,使得新的坐标系下的各个维度之间不相关,从而实现降维。
02
为什么要降维
设想一下,假如我有几个学生的英语和数学成绩,想要描述学生分数的整体情况,并将他们分入不同班级。那我也许只需要分成英语好数学差,数学差英语好以及都好都差这四个象限就行了。但是,如果我有许多学生许多门科目的成绩呢?那么情况就非常复杂了,想要用一张图描述这么多维度对三维生物来说就更不可能了。于是我就会想,要是能像只有两门那样简单就好了,这时候就要用到降维了。通过主成分分析,可以将原始数据的维度减少到k维,同时保留了最重要的特征信息。降维后的数据在一定程度上简化了计算和可视化,并且可以去除原始数据中的噪声和冗余信息。主成分分析在数据预处理、特征提取和数据可视化等领域都有广泛的应用。
03
PCA是怎么做的呢?
简单来说:
1. 将坐标轴中心移到数据的中心,然后旋转坐标轴,使得数据在C1轴上的方差最大,即全部n个数据个体在该方向上的投影最为分散。意味着更多的信息被保留下来。C1成为第一主成分。
2. 找一个C2第二主成分,使得C2与C1的协方差(相关系数)为0,以免与C1信息重叠,并且使数据在该方向的方差尽量最大。
以此类推,找到第三主成分,第四主成分……第p个主成分。p个随机变量可以有p个主成分。这些主成分是原始数据的线性组合,可以用来表示原始数据中的信息。
大家听懂了吗?没听懂也没有关系,我们直接来举个低维的例子看看具体是怎么做的吧!
首先,我们建立一个三维直角坐标系
在里面撒上一些点,当然,每个点具有三个维度的(经过标准化的)观测值。
计算每个点的每个维度的算术平均,我们得到所有点的重心(下图中红点)。
移动坐标系,使原点与重心重合。
接着,我们找到所有数据点的最优拟合直线,称为PC1,将数据点投影上去,也就是说取过每个数据点(图中为i)正交于PC1的直线与PC1的交点。
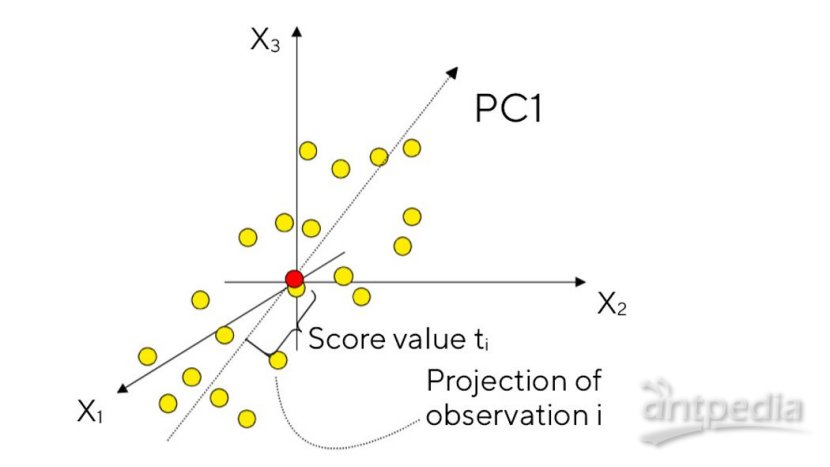
接下来再在与PC1正交的方向中找到一条最适回归线经过原点,这样就形成了一个平面(二维空间),然后通过数据点在PC1和PC2上的投影就能找到在平面上的投影。这样一来,三维数据向二维的降维就完成啦!
04
作图(Python代码实现)
下面以sklearn.datasets内置的鸢尾花数据为例,展示用python代码绘制有置信椭圆的散点图。
import numpy as npfrom matplotlib.patches import Ellipseimport pandas as pdimport matplotlib.pyplot as pltimport randomfrom sklearn.decomposition import PCAimport matplotlib.pyplot as pltfrom sklearn.datasets import load_iris''''''用于计算样本点的协方差矩阵''''''def plot_point_cov(points, nstd=3, ax=None, **kwargs): # 求所有点的均值作为置信圆的圆心 pos = points.mean(axis=0) # 计算协方差矩阵 cov = np.cov(points, rowvar=False) return plot_cov_ellipse(cov, pos, nstd, ax, **kwargs)''''''计算置信椭圆的参数''''''def plot_cov_ellipse(cov, pos, nstd=3, ax=None, **kwargs): # nstd=3代表99% 的置信区间 def eigsorted(cov): cov = np.array(cov) vals, vecs = np.linalg.eigh(cov) order = vals.argsort()[::-1] return vals[order], vecs[:, order] if ax is None: ax = plt.gca() # 计算输入协方差矩阵的特征值和特征向量并返回排好序的结果 vals, vecs = eigsorted(cov) # 计算向量 vecs 的第一列的极角,并将结果转换为角度制 theta = np.degrees(np.arctan2(*vecs[:, 0][::-1])) # 得到绘制的椭圆的宽度和高度 width, height = 2 * nstd * np.sqrt(vals) ellip = Ellipse(xy=pos, width=width, height=height, angle=theta, **kwargs) ax.add_artist(ellip) return ellip''''''画置信圆''''''def show_ellipse(X_pca, y, pca, flag=1): # 定义颜色 colors = [''tab:blue'', ''tab:orange'', ''seagreen''] regions = [''Ethiopia'', ''Somalia'', ''Kenya''] # 定义分辨率 plt.figure(dpi=300, figsize=(8, 6)) # 三分类则为3 for i in range(0, 3): pts = X_pca[y == int(i), :] new_x, new_y = X_pca[y==i, 0], X_pca[y==i, 1] plt.plot(new_x, new_y, ''.'', color=colors[i], label=regions[i], markersize=14) plot_point_cov(pts, nstd=3, alpha=0.25, color=colors[i]) # 添加坐标轴 plt.xlim(-3.5, 4.5) plt.ylim(-1.5, 1.7) plt.xticks(size=16, family=''Times New Roman'') plt.yticks(size=16, family=''Times New Roman'') font = {''family'': ''Times New Roman'', ''size'': 16} plt.xlabel(''PC1 ({} %)''.format(round(pca.explained_variance_ratio_[0] * 100, 2)), font) plt.ylabel(''PC2 ({} %)''.format(round(pca.explained_variance_ratio_[1] * 100, 2)), font) plt.legend(prop={"family": "Times New Roman", "size": 9}, loc=''upper right'') plt.show()labels = [''setosa'', ''versicolor'', ''virginica'']iris = load_iris() #读入鸢尾花数据X = iris.datay = iris.target_names[iris.target]print("y length--------", len(y))y_category = pd.Categorical(y,ordered=True,categories=[''setosa'', ''versicolor'', ''virginica''])y = y_category.codesprint(y)print(y.shape)print(type(y[0]))n_components = 2 #为了绘图方便,将PCA的主成分设为2pca = PCA(n_components=n_components) #PCA主成分分析X_pca = pca.fit_transform(X) #数据标准化show_ellipse(X_pca, y, pca)

产生的散点图如上图所示。比较重要的说明都写成注释标在代码里了,值得注意的是,在函数plot_cov_ellipse中,nstd=3表示椭圆的长度和宽度分别扩展到 3 倍标准差的大小,覆盖了大约 99.73% 的数据。这是因为在正态分布中,大约有 99.73% 的数据落在距离均值三个标准差的范围内。因此,使用 nstd=3 绘制置信椭圆通常被认为是覆盖 99% 的置信区间。如果需要95%的置信区间时可以将nstd设为2。但需要注意的是,这里所提到的百分比是基于正态分布的假设。
好了,今天关于PCA主成分分析作图的分享就到这里了,赶紧把iris换成自己的数据集试试吧!祝大家在科研道路上一帆风顺。
.
文末看点|lumingbio
上海鹿明生物科技有限公司是欧易生物旗下从事蛋白质组及代谢组质谱检测的专业质谱组学服务公司。公司建有国内第一个空间代谢组商业服务平台,深耕质谱组学检测分析,具体包括空间代谢组、双平台代谢组、靶向代谢组、TMT标记定量蛋白组、翻译后修饰蛋白组、4D-DIA蛋白组、单细胞及超微量蛋白组、空间蛋白组等。创新质谱组学平台广泛应用于机制解析、分型诊断、标志物筛选、药靶发掘等多个领域。公司并先后获得高新技术企业、上海市专精特新企业并建有院士专家工作站,自有包括tims tof pro2在内的各类大型质谱近二十台套,年服务项目超2000项。鹿明生物协助合作伙伴发表SCI论文近千篇,成功打造以硬数据、好服务为基础,以空间代谢组为特色的质谱组学检测服务公司品牌。

精彩往期推荐
震撼来袭!7大生信工具助力,发文一镜到底!
2023-05-4

【R语言】火山图作图实操教程,让你的文章火起来!
2023-04-17
【生信分析干货】多组学数据挖掘利器—云平台趋势分析指南
2023-04-12

探云指南 | 代谢组学一键化报告上新啦——“代谢”你想要的科研图我都有~
2023-03-17

07-01 英斯特朗
连载 | 药物一致性评价与粒度分析(三)07-01 欧美克仪器
【仪器百科】LS-909丨干湿二合一激光粒度分析仪07-01 欧美克仪器
标准物质解决方案 | PFASs(全氟及多氟化合物)06-29
第九期阿尔塔有约 | 环境专题【新污染物:PFAS】技术研讨会精彩回顾及提问解答06-29
“绿色技术范式”,分析化学未来发展方向——访中国分析测试协会副理事长、辽宁省分析科学研究院原院长刘成雁教授06-29 转载仪器信息网
华西医院-标准型数显脑立体定位仪、双通道体温维持仪、体式显微镜安装完成06-29 迈越生物
科鉴检测助力2家仪器企业获得首批产品可靠性认证证书06-28 科鉴检测
德国耶拿:锂电池生命周期分析解决方案06-28 德国耶拿
AI已来!生命科学本科教学如何紧跟技术浪潮06-28 Opentrons
盛瀚售后,五星级服务的秘诀是什么?06-28 SHINE
专为汽车制造商打造的柔性解决方案——实现制程控制06-28
西北工业大学-脑立体定位仪安装完成06-28 迈越生物
会议邀请 | 第九届海上检验医师论坛06-28
卓立要闻 | 创新发展ing…6月卓立“大事小情”速览06-28 光电行业都会关注
打造信任合作伙伴!2024年度卓立汉光客户满意度调查开启06-28 光电行业都会关注
如何挑选适用于三阶光学非线性的测量系统?Z扫描测量系统来助力!06-28 光电行业都会关注
招聘启事—中国科学院沈阳自动化研究所微纳光学测量表征技术课题组06-28 光电行业都会关注
谱育科技作为主要完成方 荣获2023年度国家科学技术进步一等奖和二等奖06-28 点击关注→
仪器原理丨顶空仪与吹扫捕集仪科普小知识06-28 天美色谱