高分文章的利器——翻译组测序(Ribo-seq)
2023-06-08 16:10:57, Dr.WL 上海吉凯基因医学科技股份有限公司

翻译组测序是指对与核糖体结合的正在翻译的RNA片段进行测序,来准确获取样本中所有可翻译分子(包括mRNA和其他潜在可翻译RNA分子如lncRNA、circRNA等)的信息与精确定量。
Ribo-seq (Ribosome profiling sequencing)是最常用的一种翻译组测序技术,该技术利用RNA酶消化细胞中的RNA,得到被核糖体保护的正在翻译的RNA片段(ribosome footprints, RFs),然后对这些被核糖体保护的30nt左右的RNA片段进行富集、深度测序、分析。可获得全基因组水平的蛋白翻译效率,发现新蛋白/短肽,翻译组测序可以和转录组数据联合计算RNA的翻译效率,可以蛋白组的数据联合,通过分析瞬时翻译与蛋白累积的关系,分析蛋白的降解效应;翻译组测序是连接RNA和蛋白的桥梁。翻译组测序可以研究细胞内基因翻译的水平、区域、速率等,结合转录组、小RNA测序、蛋白组等进行关联分析,可以更精确地研究转录后调控、翻译调控机制。
翻译组测序最初是在酿酒酵母中研究,后来慢慢在很多物种中都有研究,但是比较多的还是动物,人和小鼠研究最多,植物相对较少。

Nucleic Acids Research, 2018, Vol. 46

Ribo-seq 实验流程

一、消化组织/细胞,加入放线菌酮处理(主要是固定RNA-ribosome复合物);
二、使用RNA酶消化没有被核糖体保护的RNA;
三、富集被核糖体保护的正在翻译的RNA片段(ribosome footprints, RFs),30nt左右,哺乳动物大约28nt;
四、去除蛋白,去除rRNA;
五、建库+测序+分析。

实验参数 | 参数详情 |
细胞样本 | 用Cycloheximide(吉凯提供)进行预处理,再液氮速冻寄送,≥1×107 |
组织样本 | 用含Cycloheximide的PBS清洗再液氮速冻,也可直接液氮速冻,≥300mg |
测序平台 | Illumina Nova Seq 6000 |
测序模式 | SE50 |
测序数据量 | 30M reads |
数据质量 | Q20>90%,Q30>85% |
P-site预测 | 符合高低低3nt规则分布 |
片段分布 | 20-40nt,主峰在28-30nt左右 |

Ribo-seq的数据分析



Ribo-seq数据呈现经典的3nt周期性分布

ORF预测
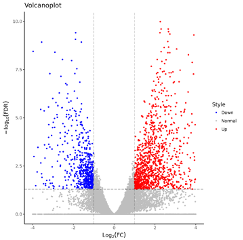
差异翻译基因火山图

差异翻译基因KEGG图

差异密码子使用频率

吉凯优势

rRNA的占比是Ribo-seq测序质量的一个关键指标,目前已发表的文章,甚至Cell上均显示rRNA占比较高,常规至少70%-80%,部分物种更是超过了90%,导致有效数据减少,数据利用效率降低,吉凯优势经过技术优化,大大降低了rRNA的占比。
样本类型 | rRNA参数 | 参数详情 |
人胃癌组织 | 40%-51% | 28-32nt |
乳腺癌细胞系 | 46%-49% | |
小鼠睾丸 | 43%-50% | |
胶质瘤干细胞 | 42%-46% | |
小鼠肝脏 | 53%-55% | |
人源胚胎干细胞 | 37%-46% | |
甲状腺癌细胞 | 37%-61% |
部分项目数据

Ribo-seq应用范围

1. 了解转录本上的核糖体分布、翻译活性;
2. 推测翻译起始位点、ORF位置;
3. 确定蛋白翻译效率;
4. 探究翻译调控和基因表达情况;
5. 和ncRNA联合,鉴定具有翻译能力的ncRNA,如:LncRNA/circRNA等。

Ribo-seq研究思路参考






1.实验技术干货
2.蛋白质组学研究
3.腺病毒简介及应用
6.单细胞测序
8.悬浮细胞专用病毒
10.测序技术研究与应用
12.腺相关病毒选择/应用
13.表观遗传研究
14.文章解析
15.国自然课题设计思路解析
16.生物信息分析及工具
17.外泌体研究
18.肿瘤免疫研究
19.高分文章
20.吉凯病毒神经方向应用案例

04-15 天美生化
兰友科技挂牌“专精特新”专板04-15
科学仪器第三方测试、可靠性工程与技术就绪度评估培训通知04-13
化繁为简 | 维生素D检测“POCT”模式新体验04-12 Chromai IVD
【邀】 科诺美与您相约沈阳“药品质量控制与检验技术大会"04-12
山海之滨话食安 | 科诺美精彩亮相第十六届食品安全技术论坛04-12
仅限2周!免疫沉淀磁珠促销来袭04-12 BEAVER
要闻丨湖南省工商联主席张健现场考察,赞赏奥谱天成为长沙打造全球研发中心城市贡献重要力量04-12
首日“出圈”!THz+ IC+ 两大生态亮相青岛市分析测试学会年会暨科仪展04-12 SHINE
国产自主分析仪器迎来重大机遇04-12
2024盖尔德纳奖重磅揭晓!新一代DNA测序、CAR-T细胞疗法获奖04-12 深究科学
【展会前沿】海能亮相德国慕尼黑分析生化博览会04-12
【展会预热】参展CBC,海能邀您共探医药全产业链新资源04-12
日立诊断,“职”“位”等你04-12 日立诊断
运用与分析的壁垒04-12 日立诊断
展会预告 | TA仪器将亮相CESC中国国际储能大会04-12 助力“双碳”的
会议邀请 | 全国化学热力学与热分析学术会议04-12
智城新品成功亮相德国慕尼黑生化展04-12 上海智城
汉化Customer Portal使用指南——供ACD/Labs软件维保期内用户04-12 ACD/Labs
虹科直播 | 4月15日起,锁定虹科智能通讯技术直播周,干货课程+现金红包大放送!04-12 虹科智能互联