蛋白研究常用数据库 | UniProt数据库介绍及使用说明
2022-05-14 05:42:51, 多组学定制服务 上海欧易生物医学科技有限公司

1
UniProt简介
目前较常用的蛋白组数据库是UniProt和NCBI这2个公共库,其中UniProt整合了Swiss-Prot、 TrEMBL 和 PIR-PSD 三个数据库,主要优势在于添加了蛋白功能注释信息。具有更新速度快、与其他数据库联系密切、分析工具齐全、使用便捷的特点,成为了目前信息最丰富、资源最广的蛋白质数据库。
一般情况下,如果蛋白质组所研究的物种已经被测序,推荐使用Uniprot数据库作为蛋白搜库匹配的数据库,如果所研究的物种在UniProt数据库中蛋白数据较少,推荐使用NCBI数据库进行搜库。
数据库链接:https://www.uniprot.org/
2
UniProt的6个主要组成的介绍
UniProt主要分为6个主要部分,该6个部分的主要介绍如下:

1. UniProtKB(Universal Protein Knowledge Base)
该部分为蛋白知识库,分为两部分,第一部分Swiss-Prot数据库,此数据库是高质量的、人工注释的、非冗余的数据库,主要来自文献报道以及E-value校验的数据。第二部分为TrEMBL数据库,该部分是计算机对大量基因组数据进行分析注释、未经人工校验的条目,其数据质量低于Swiss-Prot。
2. Supporting Data
支撑数据,主要为相关模块的说明,其包含Literature citations、Taxonomy、Keywords、Subcellular locations、Cross-referenced databases、Human diseases等。
3. UniRef(UniProt Reference Clusters)
蛋白序列参考集,分为三个数据集,分别为UniRef100、UniRef90和UniRef50,主要来自UniProtKB知识库,同时也包括UniParc归档库中部分条目。UniRef100序列将相同的序列和序列片段(来自任何生物)合并到一个UniRef条目中,用于显示代表性蛋白质的序列。
4. UNIParc (UniProt Sequence Archive)
蛋白质序列归档库,是目前数据最为齐全的非冗余蛋白质序列数据库。蛋白质可能存在于几个不同的来源数据库中,并且在同一数据库中存在多个副本。为了避免冗余,相同序列归并到同一个记录中,并赋予特定标识符(Unique Identifier,UPI)。记录包含特定标识符UPI、序列、循环冗余校验码和源数据库名称等信息。
5. Proteomes
蛋白组数据,主要是指已经完成全基因组测序物种的核酸序列翻译所得的蛋白质序列。其由测序质量较好、数据比较完整、注释比较详尽的蛋白组数据组成,但是其序列条目并非都是经过人工审阅的。
6.搜索区
搜索区可以快速找到自己感兴趣的蛋白质,检索方法可以按照蛋白质的名称、ID号、基因名、物种等。
3
UniProt搜索区域的应用
下面主要介绍3种常用检索方式的操作方法:
1.直接通过蛋白ID号搜索
(1)用浏览器登录Uniprot数据库官网:https://www.uniprot.org/;
(2)搜索框输入蛋白ID号(以P22223为例),
(3)查看P22223蛋白的生物学信息;
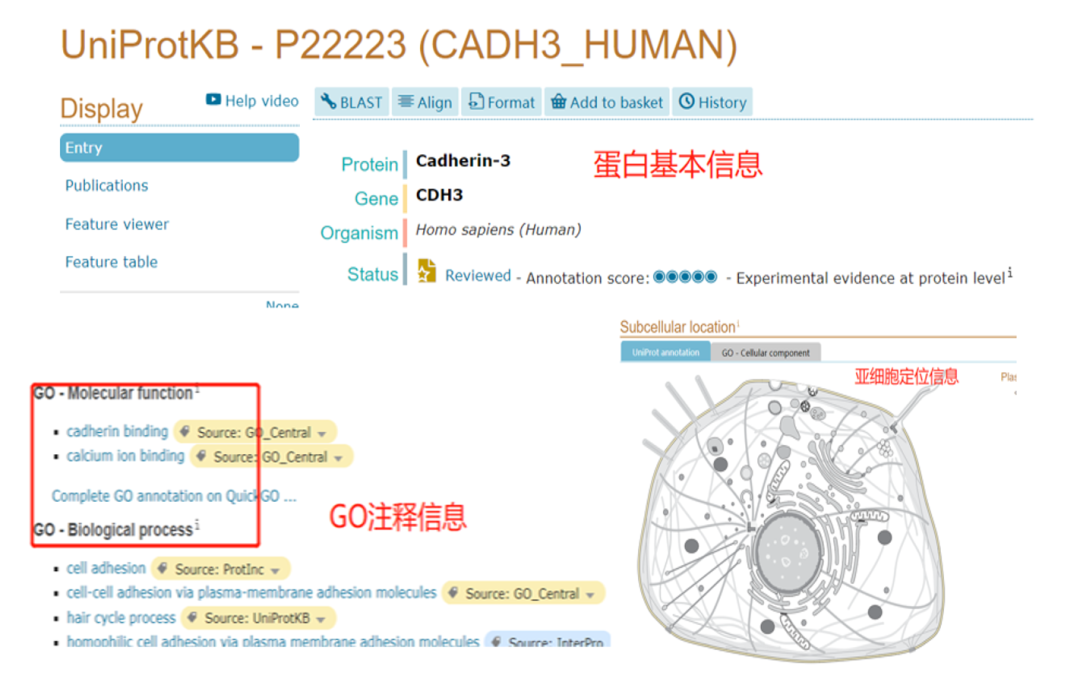
(4)下载序列数据,
2.直接通过蛋白名搜索(我们只知道蛋白名,不清楚蛋白的ID号的时候使用该搜索功能)
(1)在搜索框输入蛋白名(以Cystatin-C为例)之后search,进入下图界面;
①区域即为人物种的Cystatin-C蛋白结果。

(2)
③区域主要是蛋白名、基因名、物种信息、蛋白研究水平等
(3)红框为蛋白完整氨基酸序列,
(4)下载蛋白序列。
sp:Swiss-Prot数据库的蛋白,说明该蛋白是经过人工检查、校验的条目,是高质量的、人工注释的、非冗余的蛋白质。
P01034:蛋白在uniprot上的ID号,即蛋白的识别号(Accession号)。
CYTC_HUMAN:蛋白在uniprot上的登录名。
Cystatin-C:蛋白名称。
OS:Organism,表示物种来源,一般是物种的拉丁名,这里Homo sapiens为人的拉丁文。
GN:gene name,即基因名称。
PE:Protein Existence,即蛋白的可靠性。
SV:SequenceVersion,即序列版本号。
PE:分别有1、2、3、4、5这5个等级,数字越小可靠性越高,具体可靠性说明如下:
1. Experimental evidence at protein level 蛋白质水平验证的蛋白
2. Experimental evidence at transcript level 转录水平验证的蛋白
3. Protein inferred from homology 同源推断的蛋白
4. Protein predicted 预测的蛋白
5. Protein uncertain 未知蛋白
3.通过物种拉丁文(或者物种的编号)检索整个物种数据库。
(注:很多时候我们需要了解整个物种库的情况,此时就需要用到物种拉丁文搜索)
(1)在Uniprot数据库官网选择Proteomes子库,然后在搜索框输入物种拉丁名(以mus musculus为例,选择Organism ID为10090的小鼠为例);

(2)输入mus musculus,

(3)
如下所示:
(4)

(5)如果是序列文件,我们选择下载FASTA格式的文件(FASTA格式的文件可以直接用于数据库匹配,表格格式方便查看)。
4
Uniprot交叉引用数据库
UniProtKB条目的交叉引用部分显示了与核苷酸序列数据库、模型生物数据库、基因组和蛋白质组学资源等数据库的显式和隐式链接。一个条目可以交叉引用几十个不同的数据库,并有几百个单独的链接。如果感兴趣,直接

5总结
以上为UniProt数据库介绍及常用功能使用指南,该数据库与其他数据库资源相互联系,实现最广覆盖度、最全注释,终极目标是为科研工作者提供高质量的蛋白开源数据库,也希望本说明书能够帮助到大家。
随着上期鹿明生物推出代谢组学干货 | METLIN:一个强大的代谢物鉴定及查询的数据库后,本期的蛋白组学UniProt数据库希望能助力各位老师蛋白搜库、检索感兴趣的蛋白有所帮助。鹿明生物多年来,一直专注于生命科学和生命技术领域,是国内早期开展以蛋白组学和代谢组学为基础的多层组学整合实验与分析的团队。小鹿后期会持续推出蛋白组学及代谢组学的"库"系类文章,请各位老师持续关注... ...
重大活动关注
空代千万医学支持计划
活动超长周期:
2022.5.09~2022.8.09
深度交流:1v1技术交流探讨课题组,空间代谢组学讲座预约;
针对方向:鹿明生物空间代谢组学
申请方法:只需在线提交800字研究目的、思路及意义;即可参与活动~~

(
猜你还想看
·鹿明“库”系列
1、干货 | METLIN:一个强大的代谢物鉴定及查询的数据库
2、干货 | 临床样本数字化管理应该如何做?看完秒懂~
3、如何入局多组学实验?4D-蛋白组+代谢组双平台知识干货带您入门!
4、百万立项计划 | 突破代谢组学化合物鉴定技术瓶颈:标品库管理背后的故事
07-01 英斯特朗
连载 | 药物一致性评价与粒度分析(三)07-01 欧美克仪器
【仪器百科】LS-909丨干湿二合一激光粒度分析仪07-01 欧美克仪器
标准物质解决方案 | PFASs(全氟及多氟化合物)06-29
第九期阿尔塔有约 | 环境专题【新污染物:PFAS】技术研讨会精彩回顾及提问解答06-29
“绿色技术范式”,分析化学未来发展方向——访中国分析测试协会副理事长、辽宁省分析科学研究院原院长刘成雁教授06-29 转载仪器信息网
华西医院-标准型数显脑立体定位仪、双通道体温维持仪、体式显微镜安装完成06-29 迈越生物
科鉴检测助力2家仪器企业获得首批产品可靠性认证证书06-28 科鉴检测
德国耶拿:锂电池生命周期分析解决方案06-28 德国耶拿
AI已来!生命科学本科教学如何紧跟技术浪潮06-28 Opentrons
盛瀚售后,五星级服务的秘诀是什么?06-28 SHINE
专为汽车制造商打造的柔性解决方案——实现制程控制06-28
西北工业大学-脑立体定位仪安装完成06-28 迈越生物
会议邀请 | 第九届海上检验医师论坛06-28
卓立要闻 | 创新发展ing…6月卓立“大事小情”速览06-28 光电行业都会关注
打造信任合作伙伴!2024年度卓立汉光客户满意度调查开启06-28 光电行业都会关注
如何挑选适用于三阶光学非线性的测量系统?Z扫描测量系统来助力!06-28 光电行业都会关注
招聘启事—中国科学院沈阳自动化研究所微纳光学测量表征技术课题组06-28 光电行业都会关注
谱育科技作为主要完成方 荣获2023年度国家科学技术进步一等奖和二等奖06-28 点击关注→
仪器原理丨顶空仪与吹扫捕集仪科普小知识06-28 天美色谱