NovaSeq™ X系列与NovaSeq™ 6000结果一致性对比
2023-08-31 15:53:49 Illumina因美纳(中国)科学器材有限公司
NovaSeq™ X系列使用XLEAP-SBS化学技术,该技术是我们成熟的因美纳边合成边测序(SBS)化学技术的升级版,速度更快、保真度更高、性能更可靠。这一革新性的技术使得新一代测序仪的通量和生产力获得提升,从而改变了大规模测序的经济效益。本文中,我们展示了NovaSeq™ X系列在五大核心应用方面的提供的数据质量达到或超过了NovaSeq™ 6000基因测序仪,这些核心应用包括全基因组测序、全外显子组测序、全转录组测序、甲基化 测序和单细胞多组学测序。
01
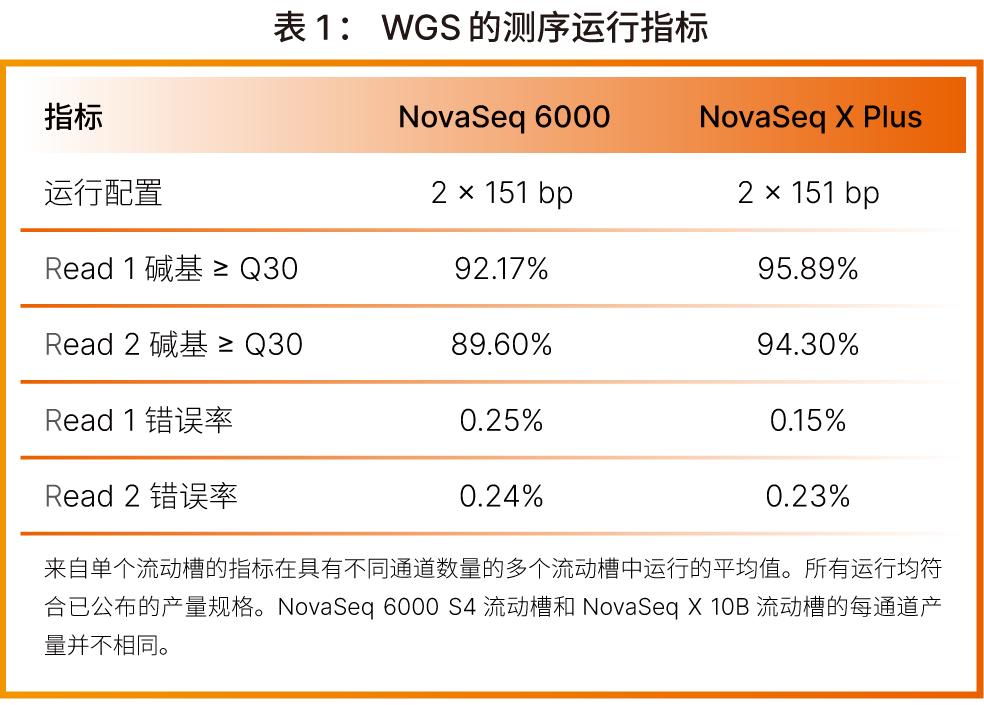
使用TruSeq PCR-Free Prep kit (因美纳:货号20015963)从NA12878 基因组 gDNA(Coriell医学研究所)样本中制备全基因组文库。使用 DRAGEN Germline pipeline v4.1 分析流程进行二级数据分析。将测序数据降采样至 30×覆盖度以比较NovaSeq X Plus和 NovaSeq 6000 基因测序仪之间的变异检出性能。
评估了全基因组测序(WGS)分析指标,包括单核苷酸变异(SNV)和片段插入缺失(INDEL)的准确率和查全率。NovaSeq X Plus和NovaSeq 6000基因测序仪均可提供高质量的数据和高度准确的变异检出(表1、表2)。这些数据表明,NovaSeq X 系列的WGS结果达到或超过了 NovaSeq 6000基因测序仪的性能。
02
使用 Illumina DNA Prep with Enrichment, (S) Tagmentation (因美纳:货号2002554)从 NA12878基因组gDNA (Coriell医学研究所)样本中制备外显子组文库,再由Twist Comprehensive Exome Panel(Twist Bioscience:货号102033)捕获其中的目标基因组区域。使用 DRAGEN Enrichment pipeline v4.0.3 分析流程进行二级数据分析。根据Genome in A Bottle(GIAB) v3.3.2 真值集和hg19-alt-aware参考基因组评估变异检出的准确度。将测序数据降采样至每个样本30M reads以比较NovaSeq X Plus和NovaSeq 6000基因测序仪之间的变异检出性能。
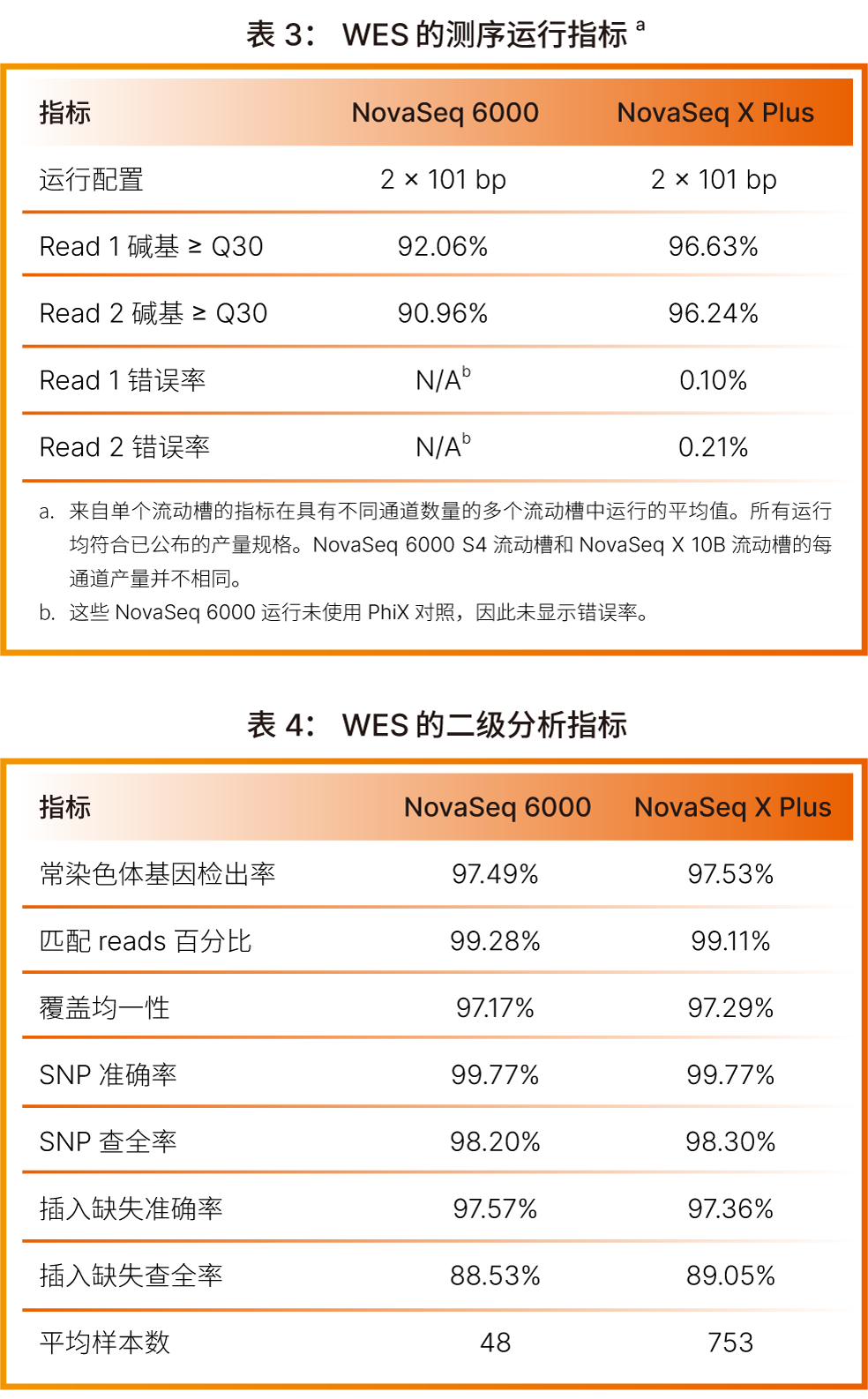
对全外显子组测序(WES)的初级和二级分析指标进行了评估,包括质量分值、错误率、常染色体基因检出率、匹配reads百分比、覆盖均一性以及 SNV和插入缺失的准确率和查全率。NovaSeq X Plus和NovaSeq 6000基因测序仪均可提供高质量的数据和高度准确的变异检出(表3、表4)。这些数据表明NovaSeq X系列的WES结果与NovaSeq 6000基因测序仪的性能相当。
03
使用 Illumina Stranded Total RNA Prep with Ribo-Zero Plus (因美纳:货号20040529)和 Illumina Stranded mRNA Prep(因美纳:货号20040534)从白血病细胞系RNA: HL-60(Thermo Fisher Scientific:货号AM7836)和K562(BioChain:货号R1255820-50)以及乳腺癌细胞系RNA: MCF7(BioChain:货号 R1255830-50)中制备总 RNA 和信使 RNA(mRNA)文库。使用 DRAGEN RNA pipeline v4.1 分析流程进行二级数据分析。所有样本的测序数据均降采样至 10M reads以比较基因表达数据。数据与基因组参照序列联盟的人类标准参考基因组 GRCh38(hg38 组装)进行比对。
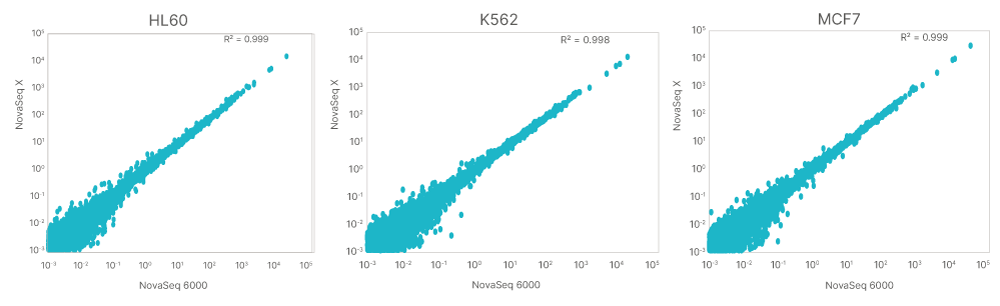
图1:全转录组总RNA-Seq 相关性一一癌细胞系: HL-60、K562和 MCF7 的每百万转录本(TPM)。

图 2:全转录组 mRNA-Seq 相关性一一癌细胞系:HL-60、K562 和 MCF7 的每百万转录本(TPM)。
NovaSeq X Plus 和NovaSeq 6000基因测序仪的总 RNA-Seq数据质量(表5)和mRNA-Seq 数据质量(表6)均高于已公布的规格。对于总RNA-Seq(图1)和mRNA-Seq(图2),两个平台的转录本定量结果具有出色的一致性(R2 > 0.99)。这些数据表明,在NovaSeq X 系列上进行全转录组测序产生的数据质量达到或超过了NovaSeq 6000基因测序仪的性能。
04
使用Zymo-Seq WGBS Library Kit(Zymo research:货号D5465),结合 Illumina DNA Prep library prep kit(因美纳:货号20060059),从匹配的人脑和脾脏样本集(Zymo Research:货号 D5018)的重复样本中制备甲基化文库(NovaSeq X Plus基因测序仪各8个重复样本,NovaSeq 6000基因测序仪各5个重复样本)。未甲基化大肠杆菌对照的添加量为0.25%,用于检测胞嘧啶的转化效率,据估计该效率高于99.5%。使用 DRAGEN Methylation pipeline 分析流程进行甲基化分析。将测序数据降采样至每个样本500M reads用于下游分析。
图3:全基因组甲基化测序——NovaSeq X Plus基因测序仪和NovaSeq 6000 基因测序仪的Zymo-Seq WGBS 结果显示 (A) 甲基化胞嘧啶百分比和 (B) 比对效率。

图4:全基因组甲基化测序的基因组覆盖度——NovaSeq X Plus 基因测序仪和NovaSeq 6000 基因测序仪的Zymo-Seq WGBS 结果显示了CpG 覆盖度分布。
对全基因组甲基化指标进行评估,从而比较NovaSeq X系列和NovaSeq 6000基因测序仪的性能。NovaSeq X Plus基因测序仪和NovaSeq 6000基因测序仪均展示了甲基化胞嘧啶 百分比的定量数据,与基于产品文档的预期一致(图3A)。在NovaSeq X Plus基因测序仪上检测到匹配文库的比对效率更高(图3B)。全基因组覆盖度分布图显示,NovaSeq X Plus基因 测序仪和NovaSeq 6000基因测序仪的结果相当,但高覆盖度 CpG(> 50×)有所增加(图4)。在文库制备过程中,亚硫酸氢盐或酶促转化会将未甲基化的胞嘧啶转化为尿嘧啶。这会导致文库不平衡,这种不平衡历来都是测序要面对的难题,并且通常需要高比例(> 5%)的 PhiX 来提高碱基多样性。在NovaSeq X系列上,仅需低比例(1%) 的PhiX即可实现高质量的测序运行(表7)。
05
Chromium Single Cell Multiome ATAC+基因表达能以单细胞分辨率联合读出基因表达和表观遗传学特征。用于单细胞RNA 测序(scRNA-Seq)和转座酶可及性染色质单细胞检测(scATAC-Seq)的样本是从AllCells公司提供的健康男性供体(年龄小于35岁)的冷冻保存人外周血单核细胞(PBMC)中联合制备的。按照10x Genomics经过验证的实验方案 —— 单细胞多组学ATAC +基因表达测序的细胞核分离(CG000365 Rev A)中的描述分离细胞核。配对的 scRNA-Seq和scATAC-Seq文库是从分离的细胞核中生成的,如 Chromium Next GEM单细胞多组学ATAC +基因表达用户指南(CG000338 Rev B)中所述。使用Cell Ranger ARC analysis pipeline v2.0(10x Genomics)进行数据分析以计算单细胞中的转录本和染色质可及性峰值。

图 5: 单细胞多组学基因表达一在NovaSeq X Plus 基因测序仪(蓝色)和 NovaSeq 6000 基因测序仪 (红色) 上测序的 scRNA-Seq文库的 t-SNE 图。

图6:单细胞多组学染色质可及性——在NovaSeq X Plus 基因测序仪(蓝色) 和NovaSeq 6000 基因测序仪 (红色) 上测序的 scATAC-Seq文库的t-SNE图。
评估了单细胞多组学检测的性能指标,包括用于测量基因表达的 scRNA-Seq和用于测量染色质可及性的scATAC-Seq。NovaSeq X Plus和NovaSeq 6000基因测序仪的数据质量超过了已公布的规格要求(表8、表9)。scRNA-Seq基因表达(图5)和scATAC-Seq染色质可及性(图6)的t-SNE图显示NovaSeq X Plus基因测序仪和NovaSeq 6000基因测序仪之间具有良好的相关性。

仅供研究使用,不得用于诊断。
.07-01 英斯特朗
连载 | 药物一致性评价与粒度分析(三)07-01 欧美克仪器
【仪器百科】LS-909丨干湿二合一激光粒度分析仪07-01 欧美克仪器
标准物质解决方案 | PFASs(全氟及多氟化合物)06-29
第九期阿尔塔有约 | 环境专题【新污染物:PFAS】技术研讨会精彩回顾及提问解答06-29
“绿色技术范式”,分析化学未来发展方向——访中国分析测试协会副理事长、辽宁省分析科学研究院原院长刘成雁教授06-29 转载仪器信息网
华西医院-标准型数显脑立体定位仪、双通道体温维持仪、体式显微镜安装完成06-29 迈越生物
科鉴检测助力2家仪器企业获得首批产品可靠性认证证书06-28 科鉴检测
德国耶拿:锂电池生命周期分析解决方案06-28 德国耶拿
AI已来!生命科学本科教学如何紧跟技术浪潮06-28 Opentrons
盛瀚售后,五星级服务的秘诀是什么?06-28 SHINE
专为汽车制造商打造的柔性解决方案——实现制程控制06-28
西北工业大学-脑立体定位仪安装完成06-28 迈越生物
会议邀请 | 第九届海上检验医师论坛06-28
卓立要闻 | 创新发展ing…6月卓立“大事小情”速览06-28 光电行业都会关注
打造信任合作伙伴!2024年度卓立汉光客户满意度调查开启06-28 光电行业都会关注
如何挑选适用于三阶光学非线性的测量系统?Z扫描测量系统来助力!06-28 光电行业都会关注
招聘启事—中国科学院沈阳自动化研究所微纳光学测量表征技术课题组06-28 光电行业都会关注
谱育科技作为主要完成方 荣获2023年度国家科学技术进步一等奖和二等奖06-28 点击关注→
仪器原理丨顶空仪与吹扫捕集仪科普小知识06-28 天美色谱