必看!99% 科研人会忽略的测序致命伤
2020-05-10 07:59:33 安捷伦科技(中国)有限公司

HiSeq X Ten 和 NovaSeq 大大提升了测序通量,但样本标签错配问题着实让人头疼。FFPE、液态活检样本的趋动变异频率实在太低,而建库环节、PCR 环节、测序本身的错误率就和变异频率差不多,如何将它们区分开?
优化的建库方案是解锁各项难题的一把钥匙,解题之前让我们先熟悉一下很多人傻傻分不清,但与上述问题直接相关的两种标签(index)——样本标签和分子标签。

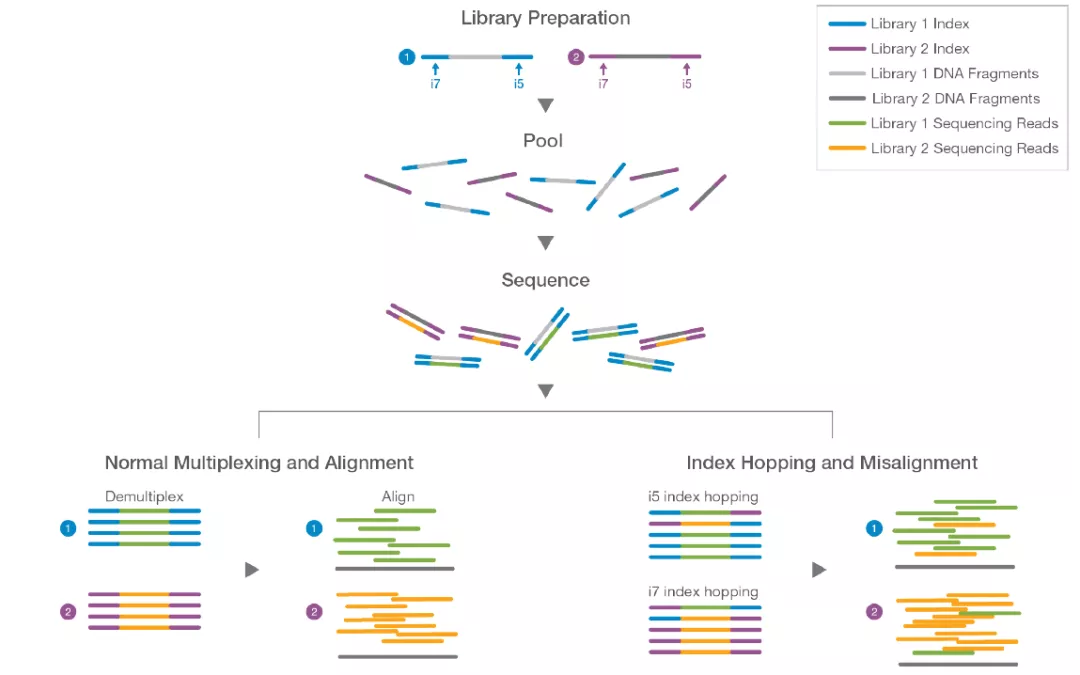

目前最普遍采用的双样本标签,相当于给样本标记加上双保险。然而组合型的双端标签(Combinational Dual Index–CDI)仍然存在标签共用的问题,以 96 CDI 为例,每一列的 i5 标签都是相同的,每一行的 i7 标签也都是相同的。当标签发生跳跃,形成新 i5 与 i7 组合时,这一组合产生的错误数据将无法被剔除。除此以外,组合型的样本标签需要将不同的 i5 与 i7 标签组合使用,一旦发生标签污染,就会引入假阳性。序列特异双端标签(Unique Dual Index–UDI)不存在标签共用问题,一对样本标签同时两两跳跃到另一段 DNA 片段上的概率几乎为零,因而可以更好的解决标签跳跃问题。





384 对序列唯一双端标签(384 UDI)满足高通量测序混样需求,同时有效应对标签错配,预混好的 384 UDI,方便操作,避免手动混合潜在的交叉污染风险 双端分子标签(Dual MBC)校正 PCR 与测序过程的假阳性,包括 PCR 早期引入的假阳性 优化的接头连接体系,大幅提高转化效率,生成高复杂度的文库,从微量的 FFEP 和液态活检样本中获得尽可能全面的信息 样本片段化的兼容性,兼容机械打断与酶切打断(后者在低频变异应用中表现更为出色) 样本的兼容性,FFPE、低质量的 FFPE, ctDNA 等样本均可采用同一操作流程 灵活的工作流程,既可以在一天内完整杂交、捕获,又可以过夜杂交 灵活的包装,包括自带磁珠的包装,方便订购 另外,安捷伦 SureSelect 酶切片段化试剂盒具有很好的 Tris/EDTA 浓度兼容性,基本无需稀释,即可对不同样本采用同一酶切程序


 长按识别二维码, 关注安捷伦视界
长按识别二维码, 关注安捷伦视界04-03 宏集科技
ICP-MS法在茶叶非金属元素分析中的应用04-03 管理员
奶茶中茶多酚含量测定方法的优化04-03 管理员
【设备更新仪器推荐】高性能全自动拉曼光谱仪——XploRA PLUS04-02 HORIBA
钙钛矿、发光材料、光电器件、电学器件、污染物去除【学术简讯24年05期】04-02 HORIBA
邀请函丨共襄2024北京国际环保展览会(CIEPEC),携手共创绿色未来04-02
赴塞上江南,启卓越之程丨2024连华科技春季技能培训大会圆满落幕04-02
智能管理 · 智慧生活 | 宏集SCADA技术线下研讨会,诚邀您参与!04-02 宏集科技
启用新一代测序化学、推出高通量流动槽,升级后的NextSeq 2000/2000-CN需要重新审视04-02 lizimo
发了Nature又发Science!全新一代10 nm空间分辨超快光谱和成像系统助力突破前沿科学04-02 Dr. Lu
JACS重要成果!超精准可调节温度控制助力钙钛矿相变的光致发光成像!04-02
使⽤Luminata提升稳定性研究效率04-02 ACD/Labs
展会预告 | 盛瀚邀您共聚青岛市分析测试学会年会系列学术报告会暨科学仪器展!04-02 SHINE
叮~基泰生物清明节放假通知,请查收!04-02 基泰生物
【内附详细日程】因科技 · 纳万物 | 2024进化发育学术论坛04-01
如何在环境监测的背景下,全面掌握三维荧光光谱?【4月16日 | 在线讲座】04-01 HORIBA
大昌华嘉开展鲁道夫(Rudolph) Autopol系列旋光仪质量服务月04-01 大昌华嘉
邀请函 | 2024中国油气开发技术年会暨油气开发新成果及新技术展示会04-01 大昌华嘉
邀请函 | IBQC第三届国际生物药质量大会04-01 大昌华嘉
邀请函 | 2024青岛市分析测试学会04-01 大昌华嘉