流程升级 | 氨探OBC组学数据批次校正平台支持SERRF归一化算法
2023-09-05 12:45:57, Untangled

一直以来,品生医疗qULTRA®多组学平台因其高灵敏度、高稳定性、高通量等优势而备受业界好评。在这背后是多组学平台在生物标志物发现、验证、应用各个环节的不断革新。
在生物标志物发现过程中,非靶向组学的数据质量的优化和提升始终是我们的核心关注点,关系到后续的差异代谢物能否被验证成功。
虽然目前学术界对于组学技术在人群队列研究中的重要性已有共识,但在实际大规模应用中仍然面临着Batch effect(批次效应)的问题。对此,品生医疗多组学平台qULTRA®在多组学数据处理环节进行了批次内/间自动校正工具OBC™(Omics Batch Correct)的不断迭代更新,以支持更大样本量、更准确、更可靠的多组学数据处理。
一、SERRF方法介绍
在大规模的非靶向代谢组和脂质组学实验中,数据集的广泛采集过程常引入各种系统误差,如批次差异、信号纵向漂移等,导致差异代谢物发现的偏差。为解决这个问题,Oliver Fiehn实验室提出了一种名为SERRF(Systematic Error Removal Using Random Forest)的归一化和批次校正方法。
SERRF是一种基于质量控制样本(QC)的归一化方法,它的基本假设是,每个变量的系统变化可以通过其他化合物的系统变化来更好地预测。这种方法选择了随机森林(RF)作为预测模型,因为RF具有以下优点:可以应用于变量多于样本的情况(p > n),适合于高通量非靶代谢与脂质组学数据的数据结构;RF可以拟合组学中经常观察到的非线性趋势;RF不受多重共线性(即变量之间的高相关性)的影响;RF能够容忍缺失值和异常值;随着树的数量的增加,RF被证明不会过拟合。
SERRF方法在处理大规模的血浆代谢与脂质组学队列研究的数据时,表现出了优秀的性能。它可以有效地利用所有相关化合物的信息来归一化每个单独的代谢物,显著减少系统误差,从而提高统计效能。
SERRF方法的实施步骤:
①自动缩放所有QC和样本的变量;②对所有变量,使用对应变量的QC强度作为响应,进样顺序、批次效应和其他代谢物的QC强度作为预测因子来训练RF模型,以拟合系统变化;③通过预测的系统误差来归一化每个化合物。
二、SERRF的实施
SERRF提供了Shiny的在线版本供免费使用,但是使用过程中发现一些问题,如速度慢、无法支持大样品数据的分析、无批次校正前后多种质控参数的评价等。为此,我们的在线批次质控和校正工具,Omics Batch Correct(https://omia.untangledbio.com/obc/ )(←直接复制并打开此链接)已经升级支持SERRF,结合OBC数据预处理和多种的质控分析评价,提供非靶向代谢组和脂质组更准确、更可靠的数据处理方案。此外,OBC基于云服务器,计算速度快,以及对SERRF算法的优化,支持大样品量的数据处理。
三、SERRF数据校正案例
我们分享的实例是1500+实验样本的血浆代谢组学数据,在分析过程中加入质控样品。QC样品为血浆样品处理后的代谢混合物,每6-10个实验随机样本质谱分析中插入一个QC样本,表征质谱检测的稳定性。PCmix组为在混合的血浆样品,与实验组一起进行样品前处理,质控样品前处理和质谱分析过程中的稳定性。代谢组学数据上传至OBC,进行质控和批次校正分析。
操作步骤
· ’Upload Data’栏——上传符合格式要求的组学数据与样本分组信息文件,可预览确认上传的数据;
· ‘Pre-process’栏——对数据进行归一化与缺失值处理操作,我们首先不归一化处理,查看原始数据质量,然后选择SERRF+Combat矫正后的查看数据改善情况。

· ‘Diagnostics’栏——查看数据,提供8种类型的数据分析结果,包括PCA,PVCA,Intensity boxplot,Molecule Intensity,RSD plot,Pearson Correlation,UMAP,TSNE。
结果展示
从图1可以看出,整体的原始数据质量很好,但在大队列样本中信号漂移不可避免,QC样本的boxplot展现了质谱检测过程中由于质谱分析造成的信号强度波动,经过SERRF归一化后,样本稳定性得到了强化,信号纵向漂移得到改善。

图1 样本中代谢物强度分布boxplot。(A)原始代谢物强度分布;(B)SERRF归一化后的代谢物强度
在原始数据PCA主成分1,2中,可以判断生物学分组明显,两类质控样本聚集但有一定分散,非生物学变异对数据有一定影响。经过SERRF校正后质控样本QC和PCmix更加聚集,生物学组内聚集度增加,组间差异更加明显。
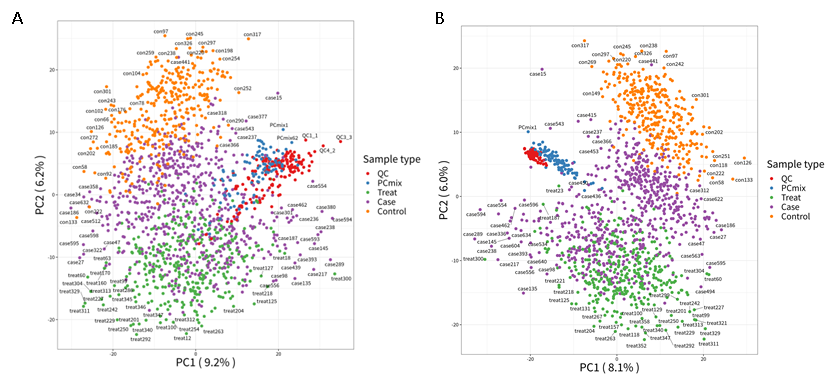
图2样本PCA分布图。(A)原始样品PCA分布;(B)SERRF归一化后的PCA分布
PVCA分析更加直观表明批次对数据的影响,原始数据中批次对数据贡献度为12.06%,批次效应有一定影响,SERRF校正后为0.02%,表明批次效应得到消除并且加强了生物学差异对数据的贡献度(图3)。
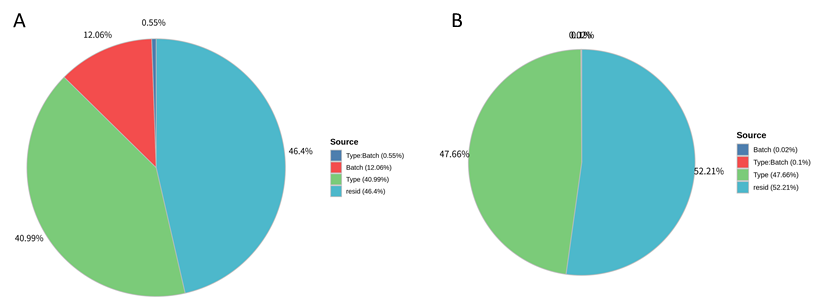
图3 PVCA结果(A,原始数据;B,SERRF归一化后)
热图聚类中Type表示生物学分组,原始结果中不同的分组有明显区分,但Batch对整个数据有一定影响,SERRF处理后改善了生物学样本的聚类,并且消除了批次聚集(图4)。

图4 样本聚类热图(A,原始数据;B,SERRF归一化后)
RSD表明了数据分组的重复情况,SERRF后减少了系统误差增加了生物学影响;各个分组生物学重复性得到增强,尤其是QC得到了极大的改善。QC样品中代谢物强度的median RSD从28.82%降低至9.57%,RSD小于30%的代谢物数目从876提高至1620(图5)。

图5 样本RSD分布(A,原始数据;B,SERRF归一化后)
在生物标志物发现过程中,非靶向组学的数据质量的优化和提升始终是我们的核心关注点,关系到后续的差异代谢物能否被验证成功。SERRF作为一种先进的归一化方法,为处理大规模的组学数据提供了新的可能性,期待在新版本的OBC工具中体验到SERRF带来的新选择,并探索其在研究中的应用价值。
1.Systematic Error Removal Using Random Forest for Normalizing Large-Scale Untargeted Lipidomics Data. Anal Chem. 2019 Mar 5;91(5):3590-3596.
2.https://slfan2013.github.io/SERRF-online/.
解构健康奥秘、探寻生命答案,氨探生物以一流的分子表型组平台和成熟的临床转化应用体系,为优秀的研究团队进行技术和数据赋能,致力于实现分子表型水平的精准诊疗。

点击阅读原文,直达OBC平台,带你探索多组学数据之美!
04-25 赛默飞生命科学
CIBF2024|H.E.L诚邀新老朋友莅临指导04-25 Don Lin
苏州佳谱科技有限公司参与制定国家标准,助力水泥窑固体废物处理技术规范发展04-25
DW行业解决方案|食品安全微生物实验室能力建设04-24 DW
全国排名公布!色谱大赛战况激烈,高手如云!04-24 市场宣传部
在众多可能中,找到你的“那一个”04-24 赛默飞生命科学
融合创新,质领未来—钢研纳克蝉联检测及科学仪器行业重磅奖项04-24 钢研纳克
4月24日直播 | 使用安捷伦Seahorse技术快速精准检测线粒体毒性04-24 安捷伦细胞分析
Nature 子刊|陆军军医大肿瘤微环境新成果,一作分享研究思路04-24 转载自生物学霸
4月25日直播 | 中国PIC/S成员资格对监管实验室的影响与含义04-24 安捷伦细胞分析
它来了,符合国家卫生行业标准的流式细胞仪性能校准服务!04-24 安捷伦科技
剑桥大学/阿斯利康利用RTCA揭示cGAS-STING 该明星分子在神经小胶质细胞炎症中的机制04-24 安捷伦细胞分析
4月26日直播 | 类器官研究大讲堂 开启精准医学新时代04-24 安捷伦细胞分析
追忆星空回响,坚守使命信念——中科科仪与“东方红一号”的峥嵘岁月04-24
激光在激光粒度分析仪中的作用04-24 真理光学粒度仪
LT3600 Plus激光粒度分析仪04-24 真理光学
真理光学诚邀你参加粉体圈第八届全国氧化铝会议04-24 真理光学粒度仪
珂睿科技诚邀您参加广西分析仪器设备应用技术交流会!04-23 珂睿marketing
终于来啦,MitoTracker小包装04-23 赛默飞生命科学
践行新质生产力:Chromeleon CDS简化您的工作流04-23 飞飞