「单细胞决策树」这一份研究攻略你必须要看!
2022-07-30 07:59:48 上海欧易生物医学科技有限公司

随着单细胞测序技术兴起,技术已日臻成熟,单细胞转录组测序凭借着可以揭示单个细胞的基因表达状态、反映细胞间异质性的优势,已经成为分子高通量测序的重要研究手段,也是当下研究的热点技术之一,可以说是科研路上的一把「利器」。但与此同时,产生的数据呈现爆发式增长,常常面对着大量的数据,熬夜熬到眼眶发黑,却仍束手无策。加上不同悬液制备方法得到的细胞/细胞核悬液不仅在数量和活性/杂质等结果表现不同,还会在某些转录信息上表现不同。「救命!我需要一份单细胞研究攻略,助我一臂之力,早日啃下这块硬骨头。」那么,欧易生物研发团队整理了详细的「单细胞决策树」,你值得拥有!让你看完直呼惊喜!
单细胞(核)悬液制备是关键步骤
温浴酶解法的双重优势:应用广泛 + 经验丰富

图 1 | 引自《Organelles | Biology for Non-Majors》;细胞外基质的主要成分(细胞外基质主要由蛋白聚糖 Proteoglycan;结构蛋白 Collagen、elastin;粘连糖蛋白 glycoprotein 构成)

图 2 | 酶解法参考网站预览(网站中收录着 30+ 种组织类型的酶解方法和对应文献)
温浴酶解后细胞转录本真实性受到质疑
低温酶解方案可「冻结」细胞转录信息
低温酶解方案使用在耐寒菌株中纯化得到的复合蛋白酶替换温浴酶解法中的蛋白酶,低温酶解过程中的 4~6 ℃ 环境温度可以抑制胞内酶的活性,降低应激应答水平。O''Flanagan C H 等人发现热解离法引入基因表达模式的改变,而低温解离法较好地维持了基因表达的稳定[3-6]。目前已被验证可以使用低温酶解法开展实验的样本类型囊括了大多数常规的组织类型。

图 3 | 38 个基因在 4 种不同条件下解离后的表达均发生了变化

图 4 | 低温酶解应用实例(酶解效果与温浴法无明显差异)
低温酶解方案可以避免批次效应
Mike Adam[4]等人在实验中发现胶原酶酶解和长酶解时间都将诱导应激基因表达,不同的酶解时间会诱导不同的表达水平,这是批次效应的因素之一。在低温酶解方案中,酶活限制了细胞应激应答,不同酶解时间之间的基因表达水平相近。这一发现表明了低温酶解可避免批次效应。

图 5 | B. Log fold changes of a 2-h vs. 30-min digestion for collagenase only as a function of log counts-per-million.
C.Log fold changes of a collagenase vs. cold protease digestion at 30-min digestion time as a function of log counts-per-million.
D. Log fold changes of a collagenase vs. cold protease digestion at 2-h digestion time as a function of log counts-per-million.
E. Log fold changes of a 2-h vs. 30-min digestion (collagenase only) compared to a collagenase vs. cold protease digestion at 2 h demonstrate a large overlap between genes affected (ρ = 0.8)
「转录抑制剂+」阻止转录发生,温浴下实现无(低)应激应答
「转录抑制剂 + 实验方案」:将转录抑制剂加入温浴酶解体系,阻断转录本的合成过程,实现温浴下的无(低)应激应答。这一技术方案目前也已经得到了验证和报道,证实了转录抑制剂的引入可以有效地「阻止」应激应答发生[7]。

图 6 | 转录抑制剂分子化学结构式
表 1 | 转录抑制剂分子作用机制


图 7 | 拟南芥叶片「转录抑制剂+」对照组与实验组差异基因的表达情况热图(S1:无抑制剂组、S2:抑制剂)
酶解法单细胞测序实验存在解离偏好性
组织中细胞外基质的多样性和细胞间连接的多样性决定了酶解法存在偏好性,例如肺组织中的非驻留免疫细胞(T、B 淋巴细胞等)在组织中结合简单,酶解过程中属于易被酶解得到的细胞,而肺泡上皮细胞结合「牢固」,要在胶原酶、弹性蛋白酶共同作用下才能得到有效酶解。解离偏好性限制了对组织全面信息的获取,2011 年,Navin[8]等开发了一种无解离偏好性的单细胞核测序方案。
单细胞核转录组测序实验(single-nucleus RNA sequencing, snRNA-seq)操作简易、无偏好、适用组织类型广泛

图 8 | 细胞膜结构示意图
单细胞核转录组测序检出免疫细胞较少
2020 年,Michal Slyper[24]等通过对多组样本单细胞核测序与单细胞测序之间的对比发现,神经母细胞瘤中,相比 SnRNA-seq,ScRNA-Seq 中免疫细胞比例更高,神经嵴、神经内分泌细胞等实质细胞大幅减少;而 SnRNA-Seq 结果中实质性细胞(尤其是恶性细胞)比例更高,但是 T 细胞比例大幅减少,B 细胞和 NK 细胞基本消失,内皮细胞、上皮细胞占比增加(图 9 左),这一发现在其余的 7 种组织类型中也得到了验证(图 9 右为乳腺癌样本)。
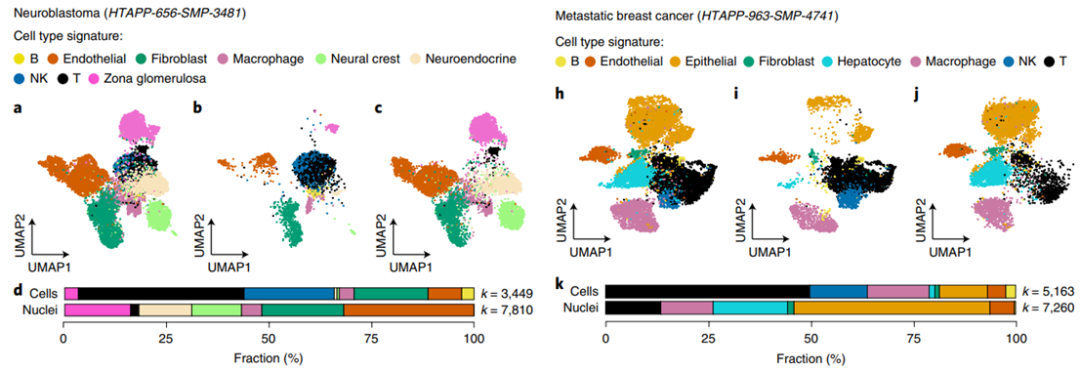
图 9 | sn-RNAseq 和 sc-RNAseq 细胞比例对比
「单细胞 + 单细胞核」实现更多的信息获取
同一组织同时开展单细胞和单细胞核测序(Sc-RNAseq + Sn-RNAseq)的实验方法是通过首先对组织全部投入进行酶解,收集易被解离的 T、B 淋巴细胞等细胞类型,紧接着对酶解后的组织进行收集,将无法得到酶解的细胞进行细胞核提取,分别进行单细胞 RNA 测序和单细胞核 RNA 测序,经过样本文库构建测试,结果是符合预期的,两个方法对同一组织展开实验得到的数据是有着互补的作用,从结果的细胞类型鉴定图示中我们可以得到,细胞核中检出的内皮细胞在细胞样本中未被检出,这一方法可以大大增加我们对一份组织中信息的了解,更多的信息也给生物信息学分析方法带来了更多的可能与机遇。在这个方案里仍然可以引入转录抑制剂的实验内容来避免应激应答对实验的影响。

图 10 | 「单细胞 + 单细胞核」转录组测序流程图

图 11 | 同一组织「单细胞 + 单细胞核」转录组测序结果(左单细胞,右单细胞核)
「exo+snRNA-seq」通讯组学助力阐释细胞相互作用
生物现象背后最根本的其实就是不同细胞之间的一个互作网络,但仅知道单个细胞的功能还不够,还缺少通讯信号。无论在正常生理还是病理过程中,外泌体都是一个最为关键的信号载体。所以我们只有把这两部分同时发掘出来,才能真正地挖掘出这个网络运作的机制。

图 12 | exo+sn-RNAseq 联合分析流程图
基于不同单细胞转录组测序产品实验特点创建的「单细胞决策树」。

图 13 | 决策树第一节点:可能性

图 14 | 决策树第二节点:科学性
图 15 | 决策树第三节点:真实性

图 16 | 决策树总览
抓住「单细胞+」机遇

END
排版人:小久
原创声明:本文由欧易生物(OEBIOTECH)学术团队报道,本文著作权归文章作者所有。欢迎个人转发及分享,未经作者的允许禁止转载。


点击“阅读全文” 收获更多精彩
08-16
文末福利 | 您的天平有福了08-16 奥豪斯
修己惠人,笃行致远!2023年卓立汉光销售培训会圆满收官08-16 光电行业都会关注
国仪量子携手清华磁共振实验室为您答疑解惑!顺磁小课堂第十九期08-16 范莹莹
手把手教学!EPR基础实验视频第一期:固体装样测试08-16 CIQTEK
手把手教学!EPR基础实验视频第二期:液体装样测试08-16 CIQTEK
培安科技:展示创新力与实力,闪耀全国石油化工行业盛会08-16 培安科技
【应用分享】西洛他唑片中有关物质的含量测定(中国药典)08-16 纳谱分析
产品推荐丨Area 背照式高灵敏光纤光谱仪08-16 oceanhood
拉曼光谱在宫颈癌转移前哨淋巴结活检中的应用08-16
百特博士讲坛第八讲,期待与您相见!08-16
Bettersize2600激光粒度分析仪08-16
走向世界的新举措——百特美国分公司正式成立!08-16 丹东百特
【百特小课堂】鞘流技术在动态图像粒度粒形分析系统中的作用08-16
气相进样口隔垫问题08-16 基泰生物
13个研发项目曝光,各大厂商在研制哪些材料物性仪器?08-16
新品上市| GF320 甲烷气体泄露检测热成像仪08-15
因科技·美丰年「农业基因组赋能计划」奖项今日揭晓!08-15
探秘赛默飞色谱质谱“0” 元购!08-15
脂质组学质谱成像技术:揭示口腔鳞状细胞癌手术边缘特征08-15 质谱创新组学
