小鼠脑组织蛋白的快速 MALDI 成像
2023-07-12 20:48:56, 质谱成像先锋队 布鲁克(北京)科技有限公司-质谱仪器


MALDI-TOF/TOF质谱因其独特的灵活和方便,被用于动物组织中的完整蛋白或蛋白原位酶切的组织成像。完整蛋白成像提供了各种蛋白异变体的信息,蛋白原位酶解成像利用TOF高分辨率和准确度以及MS/MS信息确认酶切肽。
前言
布鲁克rapifleX TOF/TOF质谱仪用于所有MALDI成像数据采集,获得分子量在2000- 24000的完整蛋白质谱图和分子量在600-2400蛋白原位酶切MS/MS谱图。空间分辨率为30μm。SCiLS™ Lab软件用于成像数据的图像处理和统计分析。
结果和讨论

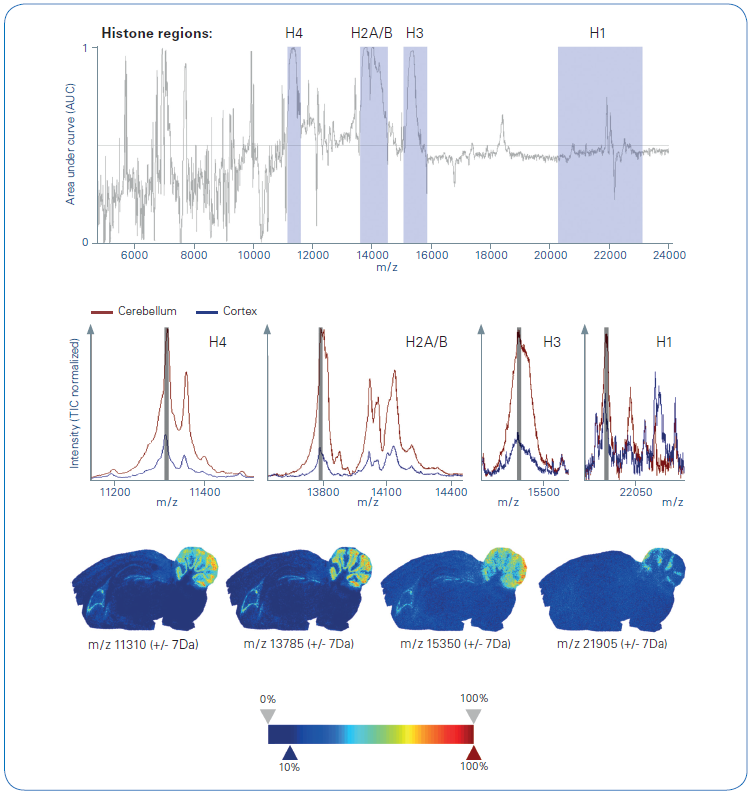
图2:完整蛋白MALDI成像的ROC分析,分割图中小脑和皮层区域的比较。ROC图, 灰色m/z范围是组蛋白区域(上),小脑(棕色)和皮层(蓝色)组蛋白放大区域的叠加平均光谱,(中), 各种组蛋白的离子图像(下)。
图3-5总结了新鲜冷冻小鼠脑切片胰酶酶切后的肽成像结果。肽谱图相似性分析给出了空间分割,显示了小鼠大脑的解剖结构(图3)。特别是分割图中红色的聚类清楚地代表小脑区域。选择MS成像中检测到的不同的肽,进行组织原位MS/MS分析,然后用MASCOT搜库得到的肽序列如图3所示。
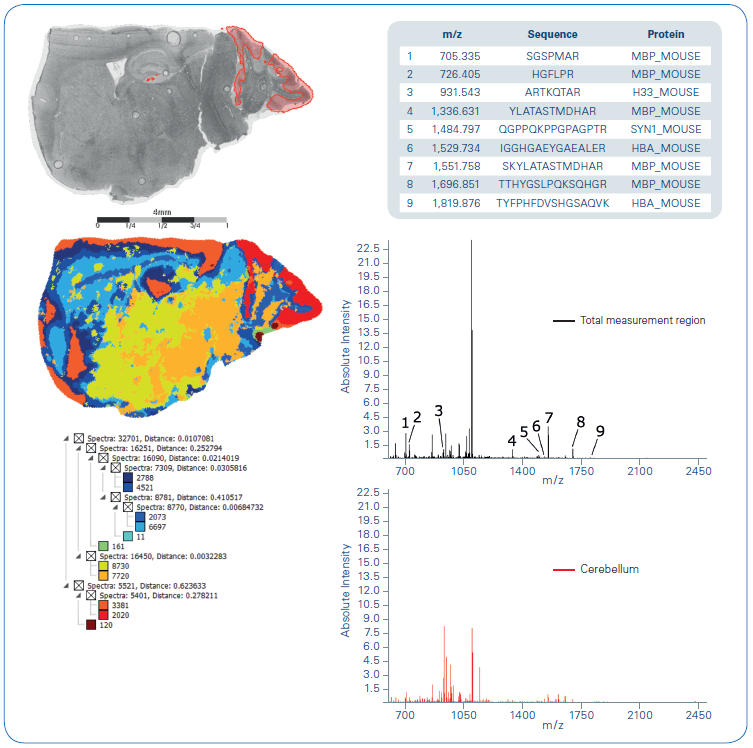

图4:胰酶切肽m/z 931.5+/-0.2Da的离子图像, 丰度最高的区域原位MS/MS用于鉴定组蛋白3.3的酶切肽序列ARTKQTAR。
其中一个被鉴定的肽是源自组蛋白3.3(Mus musculus)的ARTKQTAR m/z 931.54,图4 是它的MS离子图像,以及从最高丰度区域组织的肽原位MS/MS光谱。组蛋白3.3酶切肽在小脑区域的定位与之前描述的完整蛋白成像中观察到的H3组蛋白的空间分布高度一致。为了进一步证明这些结果,采集了酶切肽m/z 931.54的MS/MS图像,其最丰富肽碎片([b+18]7,m/z 775.4)的空间分布如图5所示。所得的MS/MS图像再次显示了组蛋白3.3酶切肽在小脑区域的明显定位,这与完整蛋白以及上述肽MS成像结果完全一致。

图5:组蛋白3.3胰酶切肽ARTKQTAR m/z 931.5的MS/MS图像,MS/MS谱图中生成MS/MS图像的最丰富的碎片离子[b+18]7(红色箭头)。
结论
布鲁克rapifleX MALDI-TOF/TOF质谱作为一个独特的多功能分析平台,能够对不同阶段的蛋白质进行MALDI组织成像, 包括Top-down完整蛋白成像和胰酶切肽MS成像,以及组织原位MS/MS肽鉴定和酶切肽MS/MS成像。这些工作流程可以提供相互补充的信息。虽然Top-down的方法在检测单个蛋白变异体的能力是独特的,但酶切肽的MS和MS/MS成像增加了额外的特异性维度,并允许通过MS/MS分辨重叠的同重肽分子。本实验对小鼠脑组织中组蛋白和其他蛋白质的空间分布,显示了rapifleX多种 MALDI成像方法的能力。结合HTX基质喷雾仪和SCiLS™ Lab分析软件,rapifleX可作为高空间分辨快速MALDI组织成像系统的首选。
参考文献
S. Rauser, C. Marquardt, B. Balluff, S.-O. Deininger, C. Albers, E. Belau, R. Hartmer, D. Sukau, K. Specht, M.P. Ebert, M. Schmitt, M. Aubele, H. Höfler, A. Walch, J. Proteome Res., 2010, 9 (4), 1854–1863
S. Lahiri, N. Sun, V. Solis-Mezarino, A. Fedisch, J. Ninkovic, A. Feuchtinger, M. Götz, A. Walch, A. Imhof, Proteomics, 2016, 16, 437-447
T. Alexandrov, M. Becker, S.-O. Deininger, G. Ernst, L. Wehder, M. Grasmair, F. von Eggeling, H. Thiele, P. Maass, J. Proteome Res., 2010, 9 (12), 6535–6546

▼▼点击“阅读原文”,查看原文
03-11
标准合集 | 129个环境空气相关标准03-11
会议通知 | 2024中药材及饮片质量安全检测技术培训交流会(亳州站)03-11
仪器租赁 | 岛津 液相色谱,月租金5586元起03-11
“爆”款来袭—SOCOREX蓝盖试剂瓶03-11
二月二龙抬头 | 福立仪器祝君万般祥瑞找上头,鸿运绵绵无尽头!03-11 福立仪器
华测、谱尼、国检等16家检测行业上市公司发布2023年业绩预告!03-10
仪器租赁 | 沃特世 液质联用,月租金52000元起03-10
BIOCHINA毫厘等您 |创造层析介质“芯”未来,新品亮相+龙年限定好礼,精彩抢鲜看!03-10
高效消灭副产物 | 基于毫厘SpreX系列,纯化双特异性抗体,错配体、片段类、聚体类轻松去除,收率加倍!03-10
GLMY创想仪器丨携光谱分析仪闪耀温州工博会03-10 国产精密仪器厂家
蔡司眼镜邀你「焕新生活能见度」03-09
GLMY创想仪器丨Elvatech伊瓦特手持式光谱仪亮相广东铸造年会03-09 国产精密仪器厂家
15家全国重点实验室,联盟!03-09
仪器租赁 | Markes 热脱附,月租金9000元起03-09
夏文斌局长一行莅临开元弘盛调研03-08
阿尔塔科技稳定同位素标记技术产业化基地建设成果系列报道之七:稳定同位素标记孔雀石绿与结晶紫03-08 阿尔塔科技
用热爱不负成长!女神节,将美丽进行到底03-08
人工智能和自动化在细胞培养过程中的优势03-08 美谷分子仪器
实验室仪器您做主——免费巡检惊喜来袭!03-08 美谷分子仪器