Molecular Plant | 双平台代谢组学联合分析揭示番茄复合体中果实多酚的途径结构和代谢调控
2021-06-02 21:58:09, 多层组学定制服务 上海欧易生物医学科技有限公司

点击上方“蓝色字体”关注我们

● 前言
德国植物系统生物学研究所Alisdair R. Fernie团队和保加利亚植物系统生物学研究所在Molecular Plant期刊发表题为“Exploiting Natural Variation in Tomato to Define Pathway Structure and Metabolic Regulation of Fruit Polyphenolics in the Lycopersicum Complex”的研究成果,该研究利用GC-MS代谢组学和LC-MS代谢组学的双平台代谢组学研究方法对Solanum lycopersicum和野生番茄品种的不同组织进行了代谢组学研究,通过结合公开发表的RNA测序数据,构建了多酚代谢的物种特异性代谢网络,建立了多酚代谢生物合成框架,注释了参与类黄酮途径代谢产物及基因表达情况。该研究揭示了番茄代谢产物的生物合成框架,为基于代谢组学辅助育种提供了理论基础。此外,番茄果实成熟期间的代谢产物变化情况也为未来代谢工程提供研究方向。

中文标题:利用番茄的自然变异确定番茄复合体中果实多酚的途径结构和代谢调控
研究对象:番茄组织
发表期刊:Molecular Plant
影响因子:12.084
发表时间:2020.07
研究单位:德国植物系统生物学研究所Alisdair R. Fernie团队和保加利亚植物系统生物学研究所
运用生物技术:UPLC-FTMS靶向代谢组学+GC-MS非靶代谢组学
● 研究背景
作物驯化,导致现在的农作物种植品种的基因多样性大大减少,然而自然遗传为作物育种提供良好的种质资源。番茄作为模式作物可和多种野生型植株进行遗传杂交,具有强大的基因遗传信息,而且作为二倍体,进行遗传相关研究相对简单。近年来,番茄已成为研究类黄酮和苯丙烷类化合物的主要模式作物,然而,对番茄成熟过程中多酚类化合物以及主要代谢产物的变化仍未进行充分的研究。
研究者们对不同属的番茄的多酚代谢物进行研究,分析其潜在的生物合成和修饰反应,构成了多酚代谢网络。利用LC-MS技术提取番茄和野生番茄不同组织(未成熟果实、成熟果实、叶、茎和根部)的代谢物,分析不同组织中特异性代谢物的种类。基于代谢组学分析,发现了7个番茄品种在果实成熟时期代谢产物发生的整体变化,紧接着采用GC-MS技术分析不同番茄的未成熟和成熟果实之间的主要代谢产物差异,并进行聚类分析,表明除了PENN品种外其他品种均可明显区分未成熟和成熟水果。随后,将MapMan转录组学分析和已知的RNA测序数据结合起来对生物合成基因进行注释,形成多酚代谢网络,从而构建完整的生物合成框架,并注释了参与类黄酮途径中修饰酶的功能。
●研究技术路线
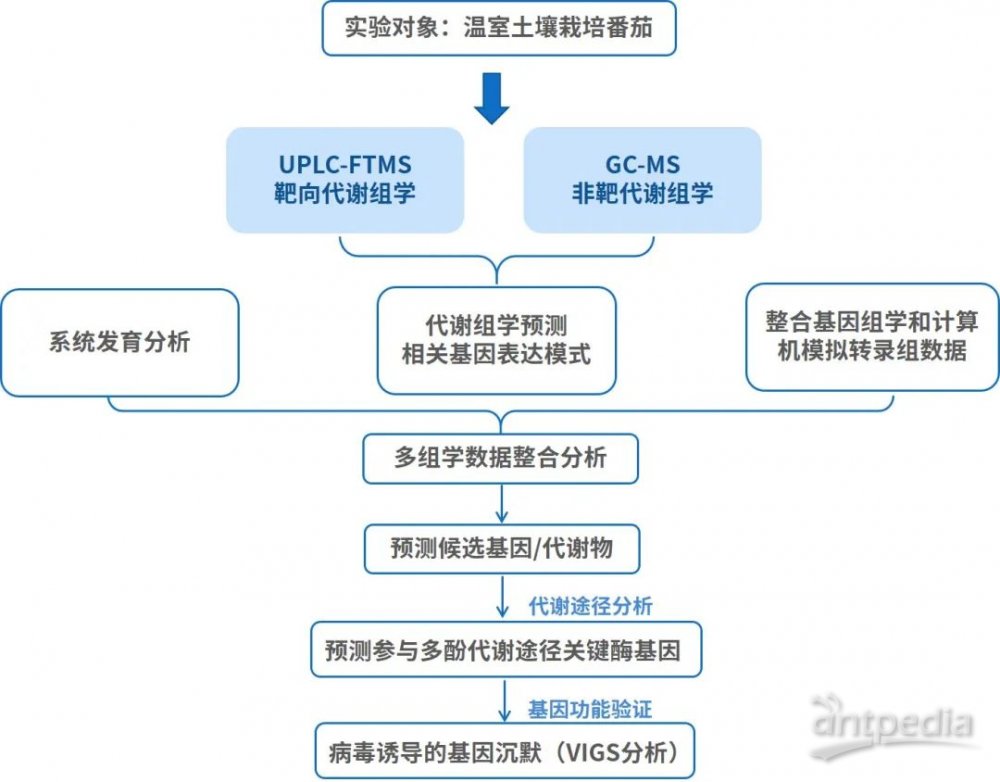
● 研究结果
1.番茄组织多酚代谢模型
作为理解番茄多酚代谢整体框架的第一步,作者通过核磁共振或通过液相色谱-质谱中的标准化合物的共洗脱来检测番茄多酚的特征(图1A)。为了评估每种代谢物的组织特异性,作者使用超高效液相色谱-傅里叶变换质谱(UPLC-FTMS)靶向代谢组学对番茄培育品种S. lycopersicum cultivar (LYCO)(M82)的未成熟果实、成熟果实、叶、茎和根(样本策略)的提取物进行了特征代谢物分析。
除根以外,在所有组织中都检测到较高水平的黄酮醇甘油三酯,但仅在果皮、叶子和茎中检测到高水平的黄酮醇单糖苷。另外,仅在果皮提取物和成熟水果中检测到查尔酮(图1B)。此外,与果实组织相比,尽管mono-CGAs在叶和茎中积累较多,但di-和tri-CGAs仅在成熟果实组织样品中被观察到(图1B)。在所有组织中都观察到羟基肉桂酸酯衍生物。图1C基于每个不同生物合成分支的主要多酚的组织特异性,对这五种组织类型进行了详细的化学分析。这些已知多酚化合物的组织特异性随后被用于分析番茄材料的自然变异,并用于番茄复合体多酚生物合成框架的重建。
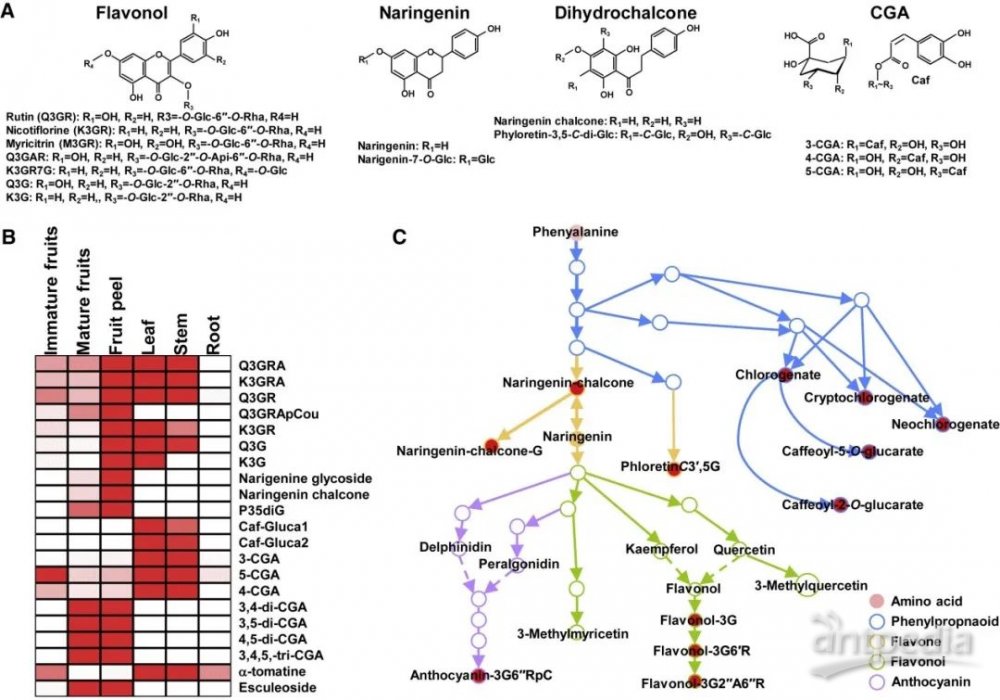
图1 | M82主要特异性代谢产物的组织特异性
2.决定番茄果实成熟的代谢组学特征
为全面了解番茄多酚的生物合成框架,包括组织/物种特异性生物合成分支,作者选择了七个主要番茄物种展开研究,包括(S. pimpinellifolium, PIMP; S. cheesmaniae, CHEE; S. chmielewskii, CHMI; S. neorickii, NEOR; S. peruvianum, PERU; S. habrochaites, HABR; and S. pennellii, PENN)(图2A)。作者对全果进行了代谢组学分析,以期了解果实成熟过程中发生的代谢变化。
为了进一步研究果实成熟过程中代谢物的变化,作者使用气相色谱-质谱GC-MS代谢组学对八种番茄的未成熟和成熟果实进行了代谢物分析(图2C)。与已知的番茄红果品种(如LYCO和PIMP)的代谢变化相比,在不同品种的果实成熟过程中观察到不同的代谢变化(图2C)。在成熟过程中,只能在LYCO, PIMP, CHEE, CHMI和NEOR中观察到β-丙氨酸和γ-氨基丁酸的减少,而除PENN以外,所有番茄材料中都可以观察到苏氨酸、谷氨酰胺和支链氨基酸(BCAAs:缬氨酸、亮氨酸和异亮氨酸)的损失。
此外,在所有绿果品种中丝氨酸和甘油酸的含量呈负相关,而奎宁酸在CHMI的成熟果实中有较高水平的积累。在水果成熟过程中,绿果品种如CHMI、PERU和PENN中苹果酸显著增加,与红色/黄色含类胡萝卜素水果中苹果酸含量的减少正好相反。苯丙氨酸是酚类代谢物的前体,在除PENN以外的所有物种中都检测到了苯丙氨酸的减少。
之后作者使用TOM2微阵列对未成熟果实样品进行转录组分析,重点是探究初级代谢基因的表达差异。
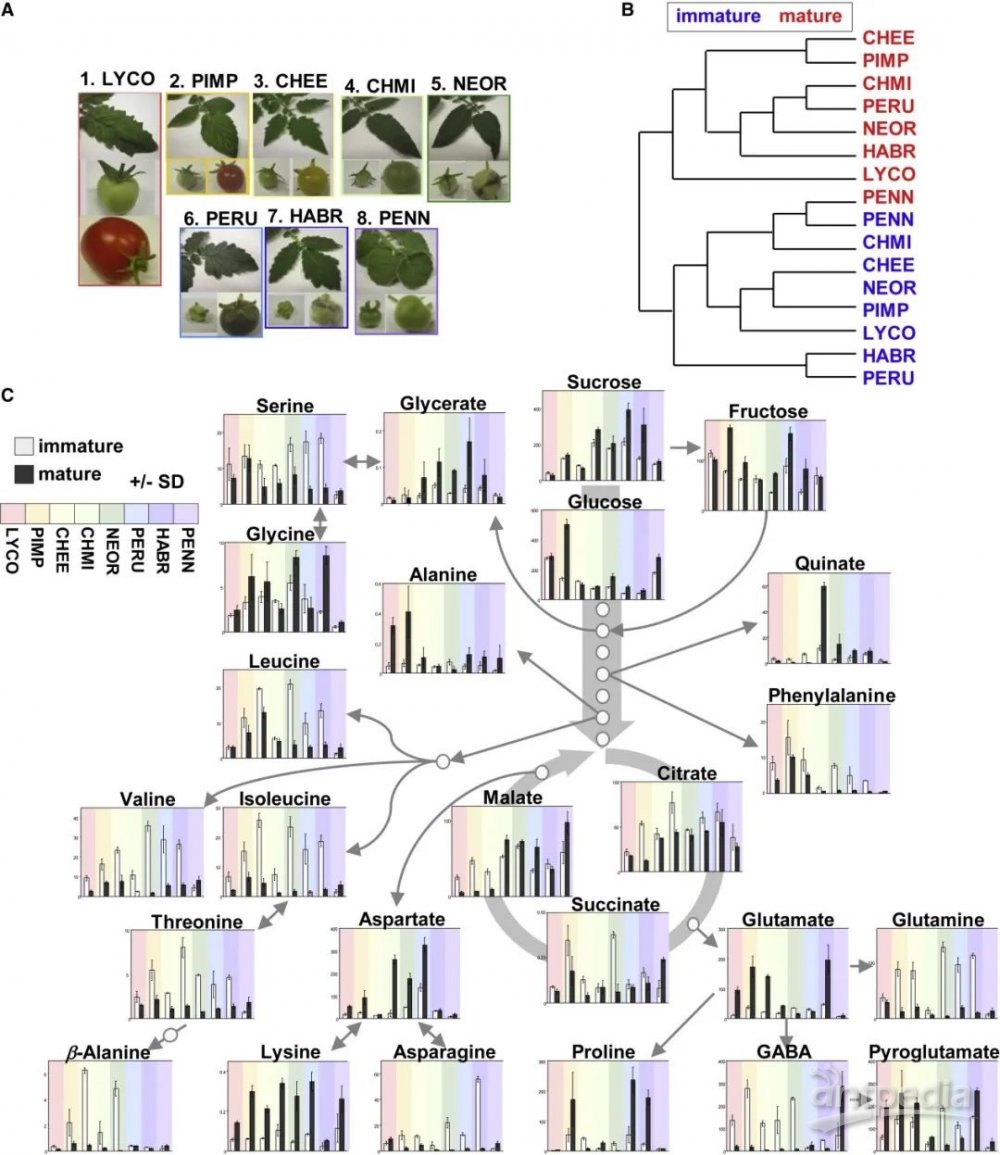
图2 | 不同番茄品种果实成熟的代谢组学特征
3.多酚代谢中物种特异性代谢多态性分析
作者对不同番茄品种的叶片、未成熟果实和成熟果实中获得的代谢物谱进行了层次聚类分析(HCA),该分析评价了单个多酚的组织特异性和种间多样性(图3)。通过对主要代谢物数据集(图2B)进行层次聚类分析(HCA),清晰地区分了未成熟和成熟的水果。根据它们的物种和组织特异性积累模式,层次聚类清晰地区分了化合物家族黄酮醇苷,假定的苯酰基黄酮醇,羟基肉桂酸盐,氯酸盐相关和二苯乙烯类。
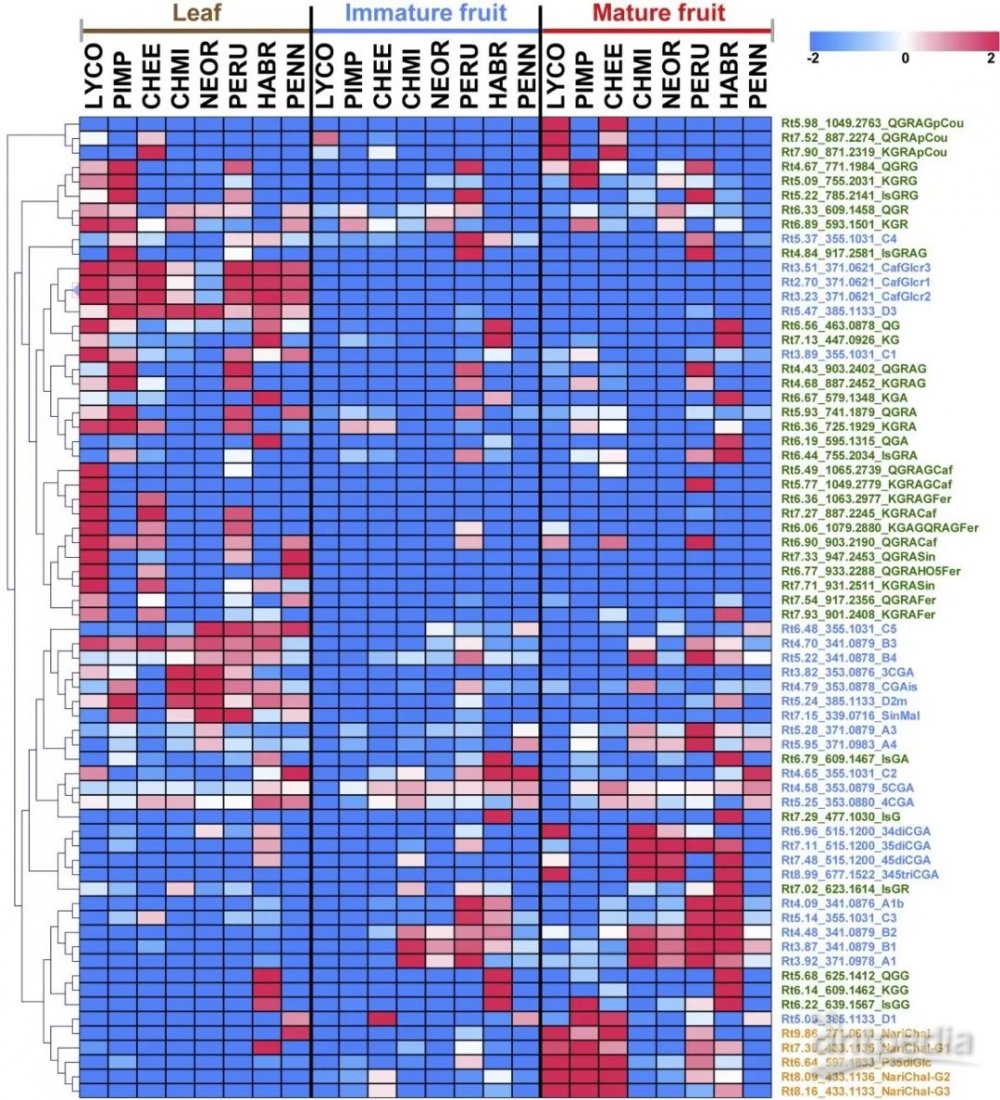
图3 | 液相色谱-质谱联用技术对番茄品种叶片、未成熟果实和成熟果实的代谢组学分析
基于不同组织或物种之间的代谢丰度和68个已鉴定推测注释的化合物的结构,作者构建了番茄多酚代谢的生物合成框架。图4汇总了多酚生物合成途径,包含有55个代谢物和37个基因的网络密度。与图1C中具有16个代谢物和28个基因的网络密度相比,可以看出这种方法在细化植物特定代谢途径方面的重要性。
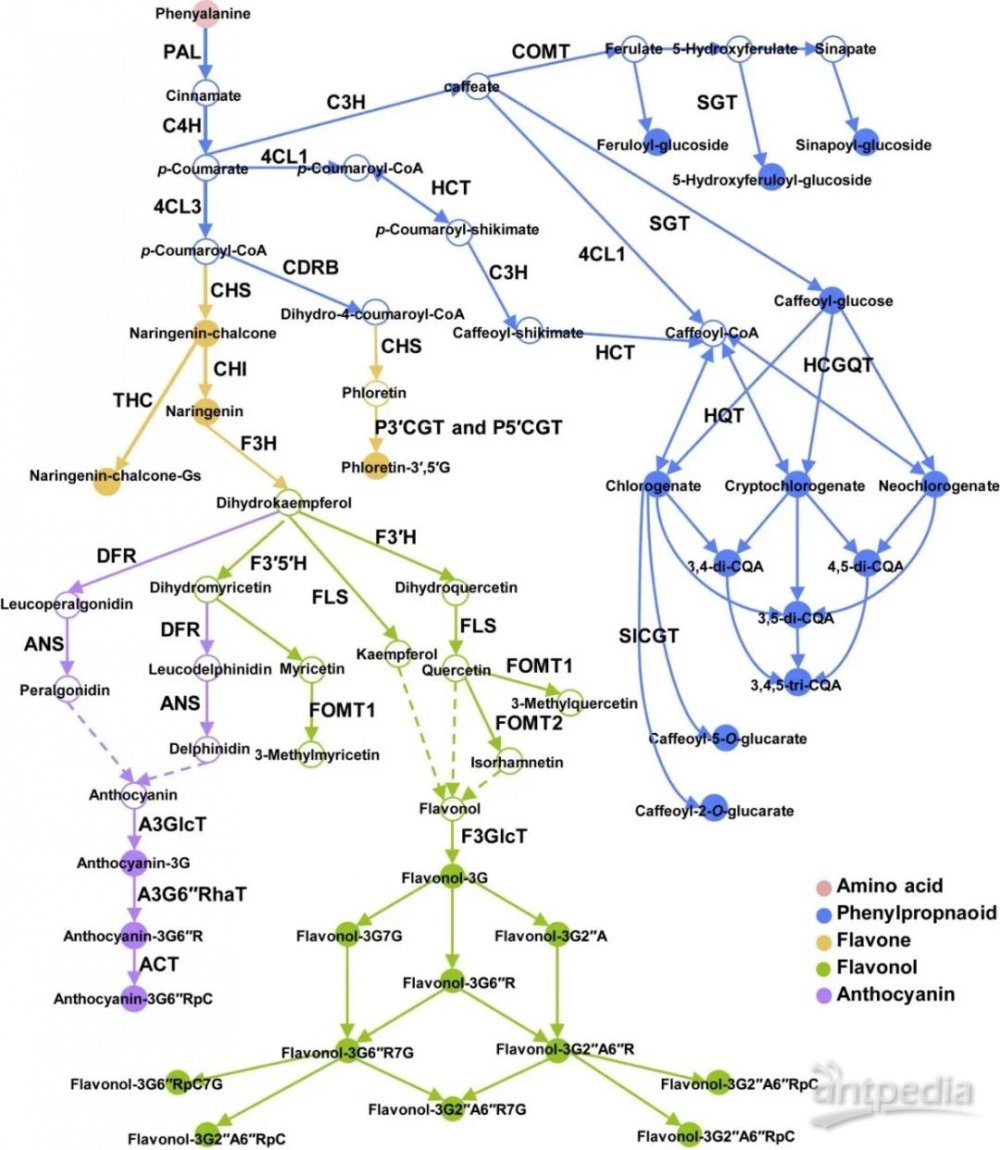
图4 | 番茄复合体的整个多酚生物合成框架概述
4.代谢组学数据预测基因表达
根据代谢物积累的存在/缺失,预测编码黄酮醇装饰酶的基因(图5A和5B)的定性转录组差异。尽管这种方法容易受到转录和代谢物数量之间潜在差异的影响,但可以对一个基因是否在一个物种或组织特异性黄酮醇生物合成分支中是活跃的做出一个初步的判断。
假设在PENN果实(图5B)和叶子中没有检测到黄酮醇-7-O-葡萄糖苷,编码黄酮醇-7-葡萄糖基转移酶的基因被预测在PENN中缺失或不表达。在黄酮醇衍生物中,结果表明一些中间体(黄酮醇-3-O-葡萄糖苷衍生物)在HABR的水果和叶子中都存在高水平的积累,表明编码具有黄酮醇-3-O-Glc-6-O-鼠李糖基转移酶活性的糖基转移酶(UGTs)的基因的表达可以忽略不计(图5B)。此外,CHMI和NEOR在果实和叶子中没有发现黄酮醇- 3Glc2''''的积累,这表明在这两个物种中编码F3G2''''ApiT的基因缺乏表达。
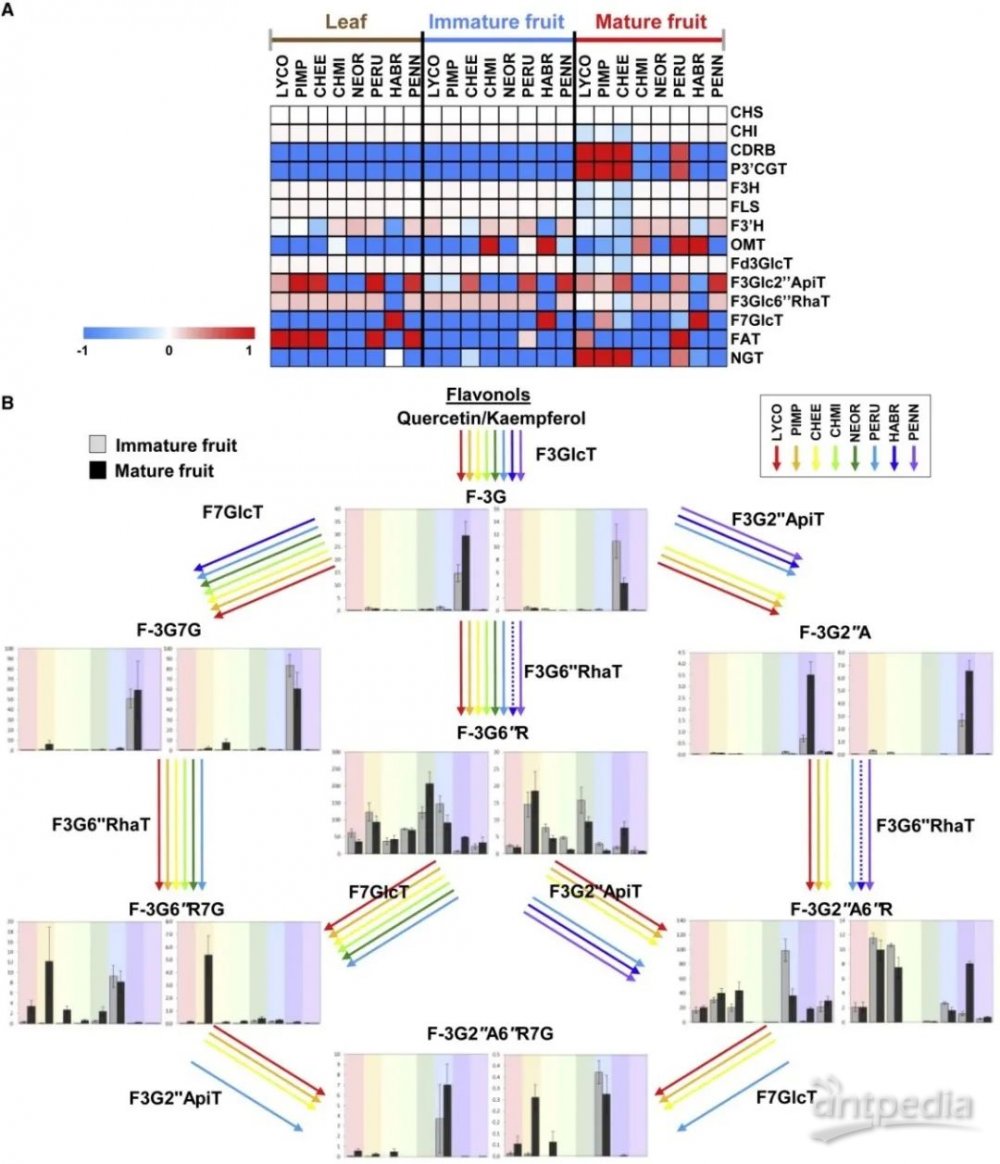
图5 | 注释生物合成框架中相关基因的预估的相对表达模式
5.类黄酮生物合成相关基因的跨物种注释
通过检索基因组的同源基因,作者将编码研究者们熟知的代谢酶的基因与作者本人提炼的多酚生物合成途径进行了匹配。通过匹配代谢物结构和酶的能力,共有26个酶基因参与类黄酮的生物合成。结果发现并表征了10个番茄物种中的类黄酮酶基因,包括:SlCHS1 (TCHS1, CAA38980, Solyc09g09151), SlCHS2 (TCHS2, CAA38981, Solyc05g053550), SlDFR, SlF3''5''H, FOMT1 和FOMT2, ScAN1, AnthOMT, SlCGT和SlFdAT1。
此外,作者对19个已知基因和另外7个推测参与番茄类黄酮生物合成途径的关键基因进行了注释。其中,推测七个关键基因(CDRB,P3''5''CGT,OMT1,Fd3GlcT,F3G2''''ApiT,Fd3G6''''RhaT,F7GlcT)参与多酚途径,现已作为催化其他植物物种中相同酶促反应的其他已知基因的直系同源物,用于类黄酮生物合成途径。
6.整合基因组学和计算机模拟转录组数据
作者通过调查番茄物种的公开可用的RNA序列数据,并将这些数据与作者的代谢数据进行交叉引用,以此来预测番茄中转录活性(代谢)基因。图6A显示了四个番茄品种(LYCO、PIMP、HABR和PENN)的叶片和两个番茄品种(LYCO和PENN)的成熟果实中类黄酮生物合成基因的计算机模拟表达分析。虽然观察到计算机转录组数据和作者预测的差异表达程度不同,但经结果表明,当显示不同的代谢表型时,代谢物谱数据可用于预测综合代谢网络中的基因表达。
为了评估参与类黄酮糖基化步骤的候选基因,作者通过系统进化树分析来自其他植物物种的编码类黄酮UDP-糖基转移酶的特征基因,比较推测了可能参与番茄类黄酮修饰的候选类黄酮UGT基因(图6B)。作者进一步测序和分析了LYCO、PIMP、NEOR、HABR和PENN品种中的类黄酮葡萄糖基转移酶的候选基因SlUGT78D-a(Solyc10g083440)(图6)。从其他番茄品种获得的UDP-糖基转移酶的活性PSPG位点高度保守。该结果支持了UGT在不同番茄物种之间的3-O-糖基化步骤中的保守作用。在HABR叶片中高表达的另一个候选基因SlUGT79B-b(Solyc10g008860)聚集在类黄酮3Glc-2''''-O-葡萄糖基转移酶附近(图6B)。
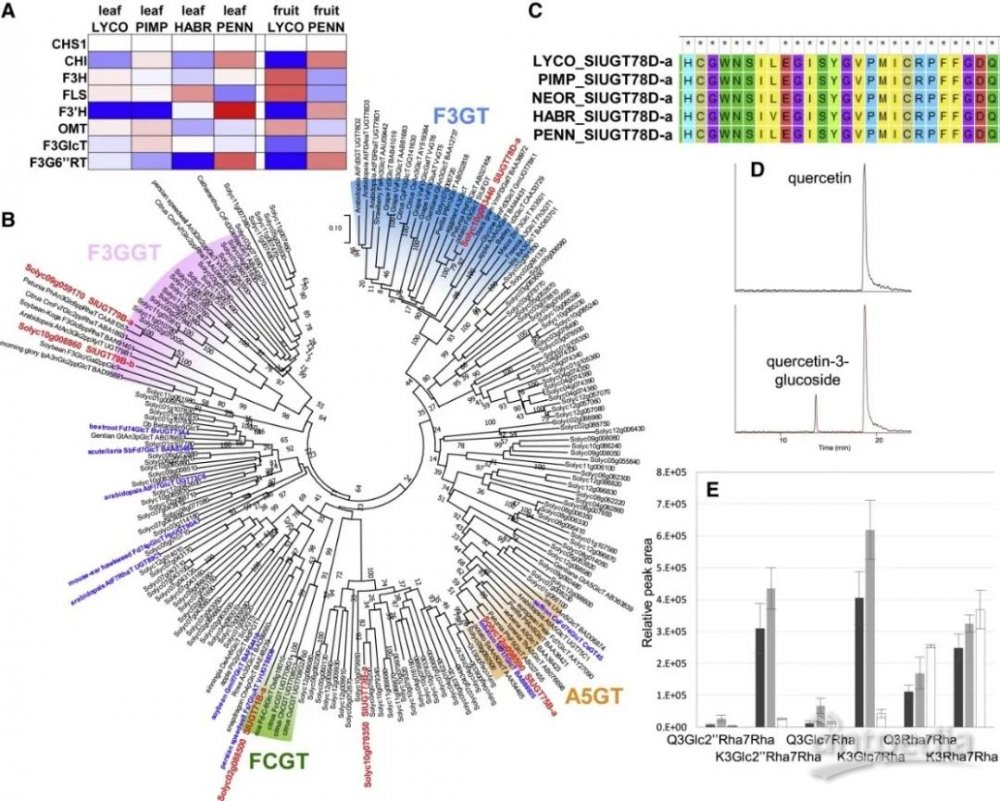
图6 | 番茄类黄酮生物合成途径中功能性UGT基因的预测
7.番茄中黄酮糖基转移酶候选基因的功能验证
作者通过使用基于转化的方法来检测UGTs的活性。为了验证标记为黄酮醇糖基转移酶的候选基因的功能,作者从LYCO中克隆了SlSolyc10g083440(SlUGT78D1-a,注释为3-O-葡萄糖基转移酶)的基因。此外,利用LYCO、PIMP、NEOR、HABR和PENN中获得的基因,研究了番茄品种间的遗传多态性(图6C)。
通过评估拟南芥,以及番茄过表达株系或抑制SlUGT78D1-a表达的番茄病毒诱导的基因沉默(VIGS)株系的代谢物图谱,从而验证了SlUGT78D1-a的基因功能。首先,作者在重组蛋白分析中检测了SlUGT78D1-a的活性(图6D)。此外,SlUGT78D1-a被转化成通常积聚类黄酮-3Glc的拟南芥(Col-0)。与野生型和拟南芥敲除AtF3GlcT(At5g17050,AtUGT78D2)的突变体相比,显示出较低的黄酮醇-3Glc积累,SlUGT78D1-a过表达的拟南芥植物显示较高的黄酮醇-3Glc相关化合物积累(图6D)。
在向未成熟果实中注射建立的基因沉默病毒后,在破果期后第10天收获成熟果实。通过LC-MS代谢组学测定果皮甲醇提取物中黄酮醇的含量,与被pTRV2空载体渗透的植物的果实中的黄酮醇相比,SlUGT78D1-a基因沉默抑制系中的黄酮醇-3-葡萄糖苷的水平明显降低。
● 相关讨论
本研究对番茄几种组织中次生代谢产物的化学多样性进行了跨物种分析,目的是了解这种多样性如何在整个番茄科植物复合体中出现和构造。通过次生代谢物种间和组织间多样性的重建,以及候选代谢基因的注释/鉴定,阐明了多酚生物合成途径的框架。
次生代谢物物质峰鉴定的复杂性是阐明代谢途径和后续候选基因功能验证的研究瓶颈。为了明确番茄果实中多酚的合成途径和代谢调控,作者采用了定性和定量代谢谱数据相结合的方法。通过代谢组学分析,在番茄物种中共检测到68种多酚代谢物。与野生番茄果实相比,驯化番茄果实中已知的主要多酚类物质的积累量和多样性较低。本研究中发现的驱动多酚合成多样化的基因可作为代谢组学辅助育种的目标因子。此外,通过番茄物种与组织间的自然变异分析获得的代谢数据是重建整个系统发育间代谢通路的唯一途径。这种方法也被用于推测基因表达模式(图5A)。
通过结合代谢组学数据与物种和组织特异性的数据,预测了由生物合成框架中的代谢通量估算的关键基因的表达模式。一般来说,主要多酚类物质的积累既不受代谢物转运系统的控制,也不受降解系统的控制。因此,这种方法可用于任何物物种,包括非模式植物。此外,将可能的酶促基因表达模式的预测与转录组数据整合在一起,从而进行了基因注释。因此,该方法为涉及植物多酚途径的关键基因的代谢组学辅助功能基因组学的新策略提供了案例研究。在系统发育背景下,代谢组学和基因表达/功能数据整合的组合策略对植物代谢研究非常有用,研究人员可以借鉴该方法对天然存在的物种和在自然界中此类化合物的水平进行代谢工程改造。
● 研究结论
本研究通过表征构成八种不同番茄多酚代谢网络的潜在生物合成和修饰反应,阐明了八种番茄复合体多酚生物合成的整个生物合成框架,并对相关代谢物和结构基因进行了详细注释,为基于代谢组学辅助育种提供帮助。此外,番茄果实成熟过程中的代谢变化为今后基于遗传的代谢工程设计提供了研究方向,并为研究其他重要植物物种中特定的代谢物的功能提供了理论依据。
小鹿推荐
本研究通过整合基于液相色谱-质谱(UPLC-FTMS)靶向代谢组学和气相色谱-质谱(GC-MS)非靶向代谢组学的双平台代谢组学,以及基于MapMan的转录组分析的交叉杂交微阵列数据和用于注释生物合成基因的公开可用的RNA测序数据,构建了完整的泛物种生物合成框架以及参与多酚代谢多样性形成的修饰酶的功能注释。这项研究推测出的生物合成框架整合方法将为目前的代谢组学辅助育种、RNA测序及遗传多态性分析提供了重要的理论基础。
.
文末看点|lumingbio
上海鹿明生物科技有限公司多年来,一直专注于生命科学和生命技术领域,是国内早期开展以蛋白组和代谢组为基础的多层组学整合实验与分析的团队。近期鹿明生物推出了4D蛋白组学&全谱代谢组联合分析
 科研福利再升级 | 新潮技术+组学联合手册—发高分必备指南免费送!包邮!
科研福利再升级 | 新潮技术+组学联合手册—发高分必备指南免费送!包邮!
欢迎长按扫码下方二维码领取4D蛋白组学&全谱代谢组联合分析电子版手册

4D蛋白组学&全谱代谢组学
长按|扫码
领取|电子版手册
参考文献:
Chen, W., Wang, W., Peng, M., Gong, L., Gao, Y., Wan, J., Wang, S., Shi, L., Zhou, B., Li, Z., et al. (2016). Comparative and parallel genome-wide association studies for metabolic and agronomic traits in cereals. Nat. Commun. 7:12767.
Fan, P., Miller, A.M., Liu, X., Jones, A.D., and Last, R.L. (2017). Evolution of a flipped pathway creates metabolic innovation in tomato trichomes through BAHD enzyme promiscuity. Nat. Commun. 8:2080.
Sulpice, R., Trenkamp, S., Steinfath, M., Usadel, B., Gibon, Y., Witucka-Wall, H., Pyl, E.T., Tschoep, H., Steinhauser, M.C., Guenther, M., et al. (2010). Network analysis of enzyme activities and metabolite levels and their relationship to biomass in a large panel of Arabidopsis accessions. Plant Cell 22:2872–2893.
Stracke, R., De Vos, R.C., Bartelniewoehner, L., Ishihara, H., Sagasser, M., Martens, S., and Weisshaar, B. (2009). Metabolomic and genetic analyses of flavonol synthesis in Arabidopsis thaliana support the in vivo involvement of leucoanthocyanidin dioxygenase. Planta 229:427–445.
Wu, S., Tohge, T., Cuadros-Inostroza, A., Tong, H., Tenenboim, H., Kooke, R., Meret, M., Keurentjes, J.B., Nikoloski, Z., Fernie, A.R., et al. (2018). Mapping the Arabidopsis metabolic landscape by untargeted metabolomics at different environmental conditions. Mol. Plant 11:118–134
猜你还想看
END
Flora 撰文
欢迎转发到朋友圈
本文系鹿明生物原创
转载请注明本文转自鹿明生物

我知道你在看哟


点“阅读原文”了解更多
04-01 质量部 戚海智
4月23日-26日,欧美克与您相约上海国际橡塑展Chinaplas04-01 欧美克仪器
【仪器百科】LS-909丨干湿二合一激光粒度分析仪04-01 欧美克仪器
【每天学点粒度知识】分析仪器的性能评价04-01
【临床研究】mNGS在神外重症中枢神经系统感染的价值研究03-30
尽揽博才英杰,共赴锦绣未来:多个职位等你加入!03-29 HORIBA
纪念梵高诞辰 —— 借光谱之力探艺术巨匠的创作精神03-29 HORIBA
设备更新指南 | 生命科学表征技术推荐03-29 HORIBA
共赴“春城之约”|第十六届中国整合生物样本学大会03-29
助力科研设备升级,推动高校设备更新 I 雷尼绍为您提供全套解决方案03-29
走进CCMT2024 | 了解雷尼绍在机床的整个生命周期中承担的角色03-29
最挑剔的流式仪器,配套的溶血素到底有多好用?03-29 市场战略中心
全域感知,生态智治|谱育科技与您相约上海环博会03-29 点击关注→
客户第一 专业专注 | 盛瀚小班培训全面提升客户专业技能03-29 SHINE
守护健康,照耀生命——HORIBA生命科学主题研讨会圆满召开03-28 HORIBA
【设备更新仪器推荐】激光粒度分析仪——LA-960V203-28 HORIBA
半导体所谭平恒团队: 范德华异质结中层间声子的层间键极化率模型 | 前沿用户报道03-28 谭平恒团队
CNS争相报道能延寿50%还能治疗肿瘤的新明星:多胺的前世今生最全梳理!03-28 麦特绘谱
绘谱学堂 | 4月10日15:00开讲《代谢组学助力心血管疾病研究》03-28 麦特绘谱
Cell Metab | 巨噬细胞的代谢支持维持结肠上皮细胞稳态03-28 麦特绘谱