|
|
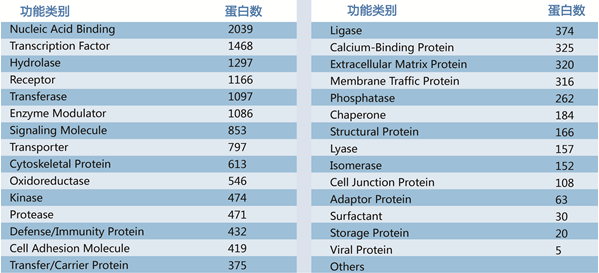
HuProt™ 20K人类蛋白质组芯片,是迄今国际zei高通量的蛋白质芯片,芯片上有19,394 种人类全长蛋白质,覆盖70%的人类基因组ORF区。重组蛋白采用酵母表达系统,逐个进行表达与纯化鉴定。在芯片上每个蛋白质均设置技术重复,并设有多种质控点,确保实验体系稳定可靠。
|
|
芯片规格:
芯片标准:1×25×76mm硅片片基,蛋白点径~60 μm,体积0.5~1 nL;
技术重复:每个蛋白点2次技术重复;
荧光检测:可使用单一或多色标签;
纯化体系:所有蛋白质在非变性条件下,通过真核酵母表达体系表达纯化;
生产条件:温度4-8℃,湿度30~40%点样,4℃过夜固定,-80℃长期保存。
蛋白类别:
HuProt™ 20K人类蛋白质组芯片通过化学修饰共价结合19275种人类蛋白质和119种小鼠蛋白质。这些蛋白广泛覆盖受体、转录因子、激酶、胞外基质蛋白、细胞骨架蛋白等30余种蛋白类型,可用于多种生物学功能的创新研究。
|
|
应用方向:
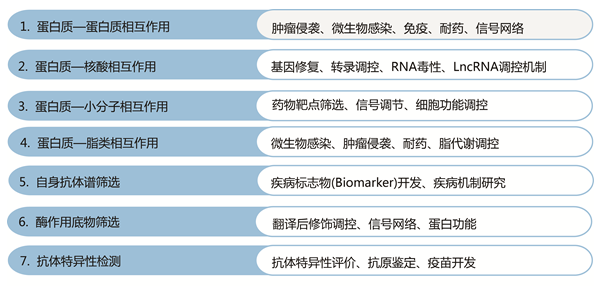
HuProt™ 20K人类蛋白质组芯片适用于以蛋白质相互作用为基础原理的各种研究领域,具备广泛的应用价值。在蛋白与蛋白相互作用筛选、蛋白与核酸相互作用鉴定方面相比传统的co-IP联合质谱鉴定的技术路线,更加高效和准确。在小分子药靶鉴定、单克隆抗体特异性筛选、脂类结合蛋白筛选、酶作用底物鉴定及自身抗体类biomarker的筛选等应用中,其技术优势不可替代。
|
|
检测原理介绍:
|
|
蛋白质相互作用谱筛选
蛋白质在体内基本全部以蛋白复合状态发挥功能,对蛋白复合物成员的识别和研究是认识和理解蛋白功能的关键。高通量HuProt™ 20K蛋白质组芯片是蛋白(抗体)互作谱鉴定的zei有力工具,通过钓饵蛋白与芯片上包被的~2万种背景信息清晰的蛋白质进行互作反应,能够极大提高相互作用蛋白的筛选范围和工作效率。
|
 |
HuProt™ 20K与免疫共沉淀(Co-IP)比较的优势
● 无需进行克隆构建、细胞培养和转染等实验;
● 无需进行免疫沉淀实验,不受靶蛋白表达量和共沉淀效率影响;
● 直接判定互作蛋白,无需进行质谱(MS)鉴定;
● 更加直观、可靠和快速,有效提高工作效率;
● 待测蛋白无特异性抗体仍然可以进行检测;
● 待测蛋白有无纯化标签(Tag)都可以检测;
● 体外实验,覆盖范围更广泛,可鉴定更多互作蛋白类型和靶点。
|
|
应用案例:HCV复制机制研究--识别宿主细胞中国HCV核心蛋白的结合蛋白(J Virol. 2013, 87(10):5718. ) 详细内容点击查看
|
|
DNA/RNA结合蛋白筛选
HuProt™20K 蛋白质组芯片可实现蛋白质与核酸(DNA/RNA)特异性结合的高通量筛选。该芯片可迅速定位与目的核酸结合的蛋白质及其背后丰富的生物信息,在转录因子筛选、LncRNA调控、 RNA毒性、病原微生物侵袭等研究中发挥重要作用。
|
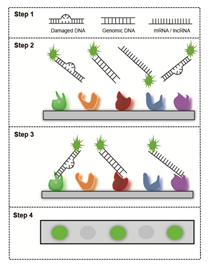 |
HuProt™ 20K 筛选核酸结合蛋白的技术优势
● 无需克隆构建、细胞培养和转染实验;
● 无需细胞核蛋白的提取,不受蛋白表达水平影响;
● 无需进行免疫沉淀实验,不受共沉淀效率影响;
● 与EMSA、ChIP、RIP比较,更适合筛选需求;
● 直接确定结合蛋白,无需进行质谱鉴定;
● 快速可靠,各蛋白信息清晰,验证实验简便;
● 体外实验,覆盖范围更广泛,可鉴定更多互作蛋白类型和靶点。
|
应用案例: lncRNA蛋白调控经典案例
详细内容点击查看 |
|
疾病标志物研究:自身抗体谱筛选
肿瘤、炎症、自身免疫性疾病(如:红斑狼疮、类风关、克隆氏病、多发性硬化)等多种疾病中,体内产生和累积大量的自身抗体。HuProt™ 20K蛋白质组芯片覆盖~20000种人类蛋白质,能够结合并大规模筛选这些人类自身抗体,从而为多种疾病的诊断、预后、药效评价等提供丰富候选标志物。
|
 |
HuProt™ 20K用于biomarker筛选的技术优势
● 芯片覆盖近2万种人类蛋白质,通量高,获得biomarker可能性大;
● 较质谱技术更适用于血清/血浆蛋白筛选;
● 特别适合自身免疫病及自身抗体类疾病标志物的筛选;
● 可追溯组织中自身抗体对应抗原的异常,理解发病机理;
● 可订制小芯片,用于扩大样本验证实验;
● 研究成果的转化应用和开发有极强便利性。
|
应用案例:人类蛋白质组芯片技术应用于移植排斥标志物筛选(Clin J Am Soc Nephro.2012;23:750–763)
详细内容点击查看 |
|
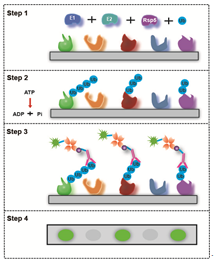
蛋白修饰与酶底物谱鉴定
HuProt™ 20K蛋白质组芯片包含的~2万种真核生物酵母表达的人类全长蛋白质,这些蛋白具备充分的生物活性, 可以为多种蛋白修饰 (如***酸化、甲基化、乙酰化、泛素化、SUMO化、NEDD化及亚***化等) 及酶促反应提供大量的候选底物,且每种蛋白生物信息背景清晰。以泛素化检测为例,如下图所示:
|
 |
HuProt™ 20K 用于酶底物谱鉴定的技术优势
● 芯片覆盖近2万种人类蛋白质,通量高,底物鉴定范围全面;
● 可进行多种修饰酶底物谱鉴定,应用范围广;
● 验证效率高,实验结果准确、全面、创新性强;
● 底物背景信息清晰,方便进行生物信息分析和深入机理解析。
|
|
应用案例:人类蛋白质组芯片技术应用于FAK激酶新底物识别(Int J Biol Sci.2015;11(4):404-410) 详细内容点击查看
|
|
小分子&单抗药物靶点筛选
化学小分子和单克隆抗体是目前zei主要的靶向药物来源,然而经典的药物研发体系往往需要15-20年的时间才能开发出适用于临床的靶向药物,且研发失败率非常高。
HuProt™ 20K 蛋白质组芯片是迄今为止zei为简便、快速和准确的药物靶点筛选实验技术,该芯片可以快速地定性和相对定量地分析经过荧光、生物素、***性同位素标记的小分子或单克隆抗体药物与~2万种人类全长蛋白质的结合情况,判断候选分子的结合靶点和特异性,为药物研发体系的改进提供强大的技术保障。
|
 |
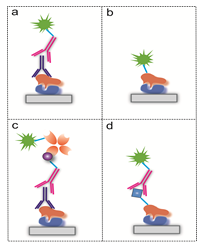
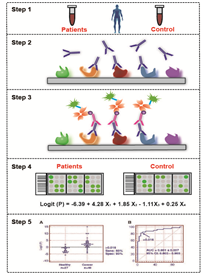
ZEB2单抗特异性研究实例展示
A.检验HuProt™ 20K 芯片质量:绿色荧光点(anti-GST),红色荧光点(Positive Control),白色方框(ZEB2蛋白);
B.竞争药厂的ZEB2单抗特异性检测结果:白色方框(ZEB2蛋白),白色箭头(抗体结合非特异蛋白);
C.本药厂的ZEB2单抗特异性检测结果:白色方框(ZEB2蛋白)。
结论:本药厂的ZEB2单抗靶点单一,较竞争药厂的单抗药物特异性更强,具有更优的应用价值。
|
| 应用案例:人类蛋白质组芯片技术应用于单克隆抗体识别研究(Mol Cell Proteomics, 2012, 11(6)) 详细内容点击查看 |
|
zei新代表文献:
蛋白质相互作用谱筛选
-
Chen W, Lin K, Zhang L, et al. The cytomegalovirus protein UL138 induces apoptosis of gastric cancer cells by binding to heat shock protein 70. Oncotarget, 2015,Dec 30(温州医科大学)
-
Fan Q, Huang L Z, Zhu X J, et al. Identification of proteins that interact with alpha A-crystallin using a human proteome microarray. Mol Vis, 2014, 20:117-124. (复旦大学附属眼耳鼻喉医院)
-
Deng R P, He X, Guo S J, et al. Global identification of O?GlcNAc transferase (OGT) interactors by a human proteome microarray and the construction of an OGT interactome. Proteomics, 2014, 14(9): 1020-1030.(上海交通大学)
-
Chen Y, Yang L N, Cheng L, et al. Bcl2-associated athanogene 3 interactome analysis reveals a new role in modulating proteasome activity. Mol Cell Proteomics, 2013, 12(10): 2804-2819. (中国科学院水生生物研究所)
-
Jung J G, et al. Notch3 Interactome Analysis Identified WWP2 as a Negative Regulator of Notch3 Signaling in Ovarian Cancer. PLoS genet, 2014, 10(10): e1004751.
-
Deng RP, et al. Global identification of O-GlcNAc transferase (OGT) interactors by a human proteome microarray and theconstruction of an OGT interactome. Proteomics. 2014;14(9):1020-30
-
Staudt N, et al. Development of an antigen microarray for high throughput monoclonal antibody selection.Biochem Biophys Res Commun. 2014; 445(4): 785-90.
-
Ma TM, et al. Serine Racemase Regulated by Binding to Stargazin and PSD-95. J Biol Chem. 2014(289): 29631-29641.
-
Chen Y, et al., Bcl2-associated athanogene 3 interactome analysis reveals a new role in modulating proteasome activity. Mol Cell Proteomics. 2013; 12(10): 2804-19.
-
Varjosalo M, et al. The protein interaction landscape of the human CMGC kinase group. Cell Rep, 2013, 3(4): 1306-1320.
DNA/RNA结合蛋白筛选
-
Howarth M M, et al. Long noncoding RNA EWSAT1-mediated gene repression facilitates Ewing sarcoma oncogenesis. J Clin Invest. 2014, 124(124 (12)): 5275-5290.
-
Kretz M, et al. Control of somatic tissue differentiation by the long non-coding RNA TINCR. Nature, 2013, 493(7431): 231-235.
-
Fan B, et al. A human proteome microarray identifies that the heterogeneous nuclear ribonucleoprotein K (hnRNP K) recognizes the 5‘ terminal sequence of the hepatitis C virus RNA. Mol Cell Proteomics. 2014; 13(1): 84-92.
-
Donnelly C J, et al. RNA toxicity from the ALS/FTD C9ORF72 expansion is mitigated by antisense intervention.Neuron, 2013, 80(2): 415-428.
自身抗体谱筛选
-
Yang L, et al. Identification of serum biomarkers for gastric cancer diagnosis using a human proteome microarray. Mol Cell Proteomics, 2015: mcp. M115. 051250.(上海交通大学)
-
Hu C, et al. Autoantibody Profiling on Human Proteome Microarray for Biomarker Discovery in Cerebrospinal Fluid and Sera of Neuropsychiatric Lupus. PLoS One. 2015 May 8;10(5):e0126643. (北京协和医院)
-
Delville M, et al. A circulating antibody panel for pretransplant prediction of FSGS recurrence after kidney transplantation. Sci Transl Med, 2014, 6(256): 256ra136-256ra136.
-
Bonsignori M, et al. An autoreactive antibody from an SLE/HIV-1individual broadly neutralizes HIV-1. J Clin Invest. 2014, 124(4): 1835.
-
Taguchi A, Taylor AD, Rodriguez J, et al. A Search for Novel Cancer/Testis Antigens in Lung Cancer Identifies VCX/Y Genes, Expanding the Repertoire of Potential Immunotherapeutic Targets. Cancer Res, 2014, 74(17): 4694-4705.
-
Yang G, Holl T M, Liu Y, et al. Identification of autoantigens recognized by the 2F5 and 4E10 broadly neutralizing HIV-1 antibodies. J Exp Med, 2013, 210(2): 241-256.
-
Mias GI, et al. Specific plasma autoantibody reactivity in myelodysplastic syndromes. Sci Rep. 2013; 3: 3311.
-
Charpin C, et al. New autoantibodies in early rheumatoid arthritis. Arthritis Res Ther, 2013, 15(4): R78.
-
Hu C J, et al. Identification of new autoantigens for primary biliary cirrhosis using human proteome microarrays. Mol Cell Proteomics, 2012, 11(9): 669-680. (北京协和医院)
-
Zingaretti C, et al. Identification of new autoantigens by protein array indicates a role for IL4 neutralization in autoimmune hepatitis. Mol Cell Proteomics. 2012; 11(12): 1885-97.
-
Sigdel TK, et al. Non-HLA antibodies to immunogenic epitopes predict the evolution of chronic renal allograft injury. J Am Soc Nephrol. 2012; 23(4): 750-63.
蛋白修饰与酶底物谱鉴定
-
Dwyer S F, et al. Identification of Novel Focal Adhesion Kinase Substrates: Role for FAK in NFκB Signaling. Int J Biol SCI. 2015, 11(4): 404.
-
Lee Y I, Giovinazzo D, Kang H C, et al. Protein microarray characterization of the S-nitrosoproteome. Mol Cell Proteomics, 2014, 13(1): 63-72.
-
Moore K E, et al. A general molecular affinity strategy for global detection and proteomic analysis of lysine methylation. Mol cell, 2013, 50(3): 444-456.
-
Feijs K L, et al. ARTD10 substrate identification on protein microarrays: regulation of GSK3beta by mono-ADP-ribosylation. Cell Commun Signal, 2013, 11(5).
-
Radu M, Rawat S J, Beeser A, et al. ArhGAP15, a Rac-specific GTPase-activating protein, plays a dual role in inhibiting small GTPase signaling. J Biol Chem, 2013, 288(29): 21117-21125.
-
Tarrant M K, Rho H S, Xie Z, et al. Regulation of CK2 by phosphorylation and O-GlcNAcylation revealed by semisynthesis. Nat Chem Biol. 2012, 8(3): 262-269.
小分子&单抗药物靶点筛选
-
Zhang H, et al. Systematic identification of arsenic-binding proteins reveals that hexokinase-2 is inhibited by arsenic. PNAS, 2015: 201521316.(上海交通大学)
-
Liu, S et al. Characterization of monoclonal antibody‘s binding kinetics using oblique-incidence reflectivity difference approach.Taylor & Francis.2015; 7:110-119.
-
Jeong J S, et al. Rapid identification of monospecific monoclonal antibodies using a human proteome microarray. Molecular & Cellular Proteomics, 2012, 11(6): O111. 016253.
-
To C, et al. Synthetic triterpenoids target the Arp2/3 complex and inhibit branched actin polymerization. J Biol Chem. 2010; 285(36): 27944-57.
|