400-6699-117转1000
咨询列表
北京诺禾致源科技股份有限公司
您好,欢迎您查看分析测试百科网,请问有什么帮助您的?



| 参考报价: | 面议 | 型号: | 全基因组测序 |
| 品牌: | 诺禾致源 | 产地: | 北京 |
| 关注度: | 150 | 信息完整度: | |
| 样本: | 典型用户: | 暂无 |
400-6699-117转1000
全面挖掘DNA水平的遗传变异
利用Illumina测序平台对人类不同个体或群体进行全基因组测序,并在个体或群体水平上进行生物信息分析。可全面挖掘基因组的SNP/InDel/CNV/SV等各类变异,为筛选疾病的致病及易感基因, 研究发病及遗传机制提供重要信息。
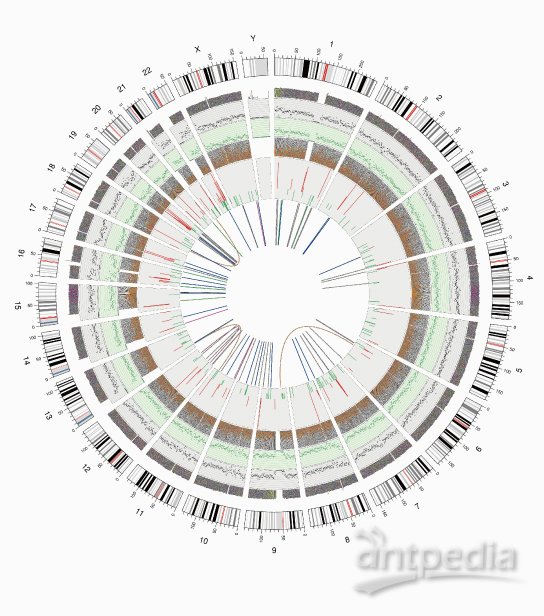
高质量,准定位,强分析
全基因组重测序可全面扫描基因组上的变异信息,一次性挖掘大量的生物标记物,该技术准确性高、可重复性好、定位精确,已被广泛应用于疾病、癌症的基因组研究。
诺禾致源独有的生物信息分析流程专注于功能性解读与多层次的变异致病性分析,并依托诺禾正常人群基因组数据库、病种数据库及药物基因组学数据库,满足多样化的科研需求。

科学的方案,高精尖的技术
从材料选取,建库测序,到数据分析,
每一步都需要科学、缜密的设计,以保障高质量研究成果。

信息分析
全基因组重测序通过对个体进行全基因组测序,全面解读基因组上的变异信息,预测该变异信息与疾病的关联性。
| 疾病基因组学 | |||
| 标准信息分析 | 高级信息分析(单基因病) | 高级信息分析(复杂疾病) | 个性化分析 |
1.数据质控:去除接头污染和低质量数据 | (一)基于变异有害性的筛选 | (一)基于变异有害性的筛选 | 1. 药物效应多态性的遗传学机理研究 |
| 癌症基因组学 | |||
| 基本信息分析 A | 基本信息分析 A+ | 高级信息分析 | 个性化分析 |
1. 数据质控 : 去除接头污染和低质量数据 | 基本信息分析 A 基础上增加5项分析内容 : | 1. MRT 高频突变基因相关性分析 | 1. 肿瘤进化树分析 |
悦读高质量测序数据,尽享HPC澎湃动力
全基因组重测序采用先进的Illumina测序平台,快速、高效地读取高质量的测序数据。
诺禾致源高性能计算平台(High Performance Computing,HPC)采用DELL计算节点和Isilon存储的高效组合,实现快速稳定的测序数据分析及交付。随着公司业务的发展,高性能计算平台将会持续更新并扩容,以保证高效的数据处理和安全的数据存储。

出色完成每一个项目环节,助力科学研究
诺禾致源疾病基因组事业部和癌症基因组事业部,致力于精准医学的科研服务。
结合丰富的项目经验,专业的项目方案指导和分析流程,保证项目准确并且快速地进行。

全基因组测序信息由北京诺禾致源科技股份有限公司为您提供,如您想了解更多关于全基因组测序报价、型号、参数等信息,欢迎来电或留言咨询。
注:该产品未在中华人民共和国食品药品监督管理部门申请医疗器械注册和备案,不可用于临床诊断或治疗等相关用途