
上海欧易生物医学科技有限公司
诚信认证:
工商注册信息已核实! 扫一扫即可访问手机版展台
扫一扫即可访问手机版展台
干货 | miRNA建库方法大放送!哪一种是你的pick?
在各种体液中的循环miRNA已经成为一类潜在的临床生物标志物。为了识别疾病特异的miRNA,small RNA seq 是一个重要的研究方法,它不但具有高效的筛选能力、特异性和灵敏性,还能够量化miRNA异构体、检测出新的miRNA等优点。尽管small RNA seq有很多的优点,但在small RNA seq的流程中也存在一定的局限性。
经典的small RNA文库构建方法基于在miRNA的3’和5’端连接两个测序接头(Norgen、Lexogen和QIAseq的建库方法),但是由于不平衡的连接效率会导致真实的miRNA水平产生严重的定量偏倚。为改进miRNA定量的准确性,发展出了3种miRNA的建库方法。
第一种方法是使用带随机核苷酸片段的接头去提高接头的连接效率。(NEXTflex的方法)。
第二种方法是无需接头连接,而是利用添加ployA并在反转录过程中进行模版转换(SMARTer的方法)。
第三种方法是依靠连接一个单端的3’接头,然后进行环化(RealSeq的方法)。
此外,在文库构建的PCR扩增过程中也会增加定量的偏倚。为了降低PCR过程中的PCR偏倚,UMI被引进用于识别和移除PCR重复(QIAseq的方法),但是这种方法在small RNA-seq中的效率是存疑的。此外,EdgeSeq平台利用捕获杂交探针和目的序列进行数据的读取,是一个在临床中易用的设计,也是small RNA-seq的一种有效的改进。本文选择了7个代表当前所有有效技术方法的small RNA-seq文库构建方法,比较了在人血浆样品中miRNA的定量结果。
实验方法

图1A:实验设计
本文选用3个健康人的血浆样品进行RNA的抽提,合并混合后进行7个不同方法的建库流程及测序分析。同时以miRXplore 通用参照作为阳性参照进行平行实验,它包含950多个等摩尔比合成的miRNA。每种方法均设计技术重复。建库方法流程及循环数根据各自试剂流程进行。
实验结果
2.1 方法间的相关性比较

图1B:miRXplore 和 plasma样品在不同方法间的相关性热图
相关性数据表明每个方法的结果在方法内都具有较高的相关性,而在方法间则相关性均较差。表明每种方法均会产生特异的技术偏倚。
2.2 不同方法的结果偏倚比较

图1C: miRXplore样品的准确度。密度图表明了检测数据与预期数据间的差异倍数的分布。虚线表示期望值的两倍差异,数字表示在两倍差异内外miRNA的百分比。
因为miRXplore 样品中的miRNA数据是已知的,通过对miRXplore 样品结果进行了各方法偏倚程度的比较(图1C)。结果表明EdgeSeq 和 SMARTer的方法有最小的数据偏倚,而Norgen 和Lexogen 的方法结果偏倚最大。
2.3 偏倚因素分析

图1D: QIAseq 数据的偏差方差百分比
以往的研究表明接头连接是造成结果偏倚的重要原因,PCR过程中产生的偏倚也是有争论的。本文利用QIAseq的数据对miRXplore样品进行了所有结果偏倚因素的分析(图1D),因为QIAseq利用了UMI,能够从其他因素中分离出PCR的偏倚影响。
结果表明连接偏倚的影响占比是最高的,而PCR引起的偏倚在整体上是可以忽略不记的。这个结果也与前面的结论一致(图1C),无需接头连接的EdgeSeq 和 SMARTer的方法有最小的偏倚,而基于连接的方法偏倚最大。
为了研究导致偏倚产生的原因,对miRXplore样品中所有种类miRNA的特征进行了分析(图1E)。在RealSeq 和 SMARTer方法中,miRNA序列中的第一个核酸有较高的影响,分别贡献了44%和25%的变异性。在基于双端接头连接的方法Lexogen、Norgen和QIAseq中,miRNA的最后一个碱基和接头结构的自由能对偏倚产生影响。总之,这些结果表明连接是small RNA-seq过程中数据偏倚产生的最大来源,它能够被技术特异性及其他较多复杂因素影响。

图1E: miRXplore 样品中QIAseq 数据miRNA测序特征的方差百分比
2.4 RT-qPCR验证相关性
作者的数据也表明每种方法的的miRNA 结果都会产生各自的独特数据偏倚,然而这些结果是基于平均混合的miRNA的结果,并不能完全代表生物样品中的真实情况,因此作者在血浆样品中挑选了19个miRNA进行RT-qPCR,分析了与RNA-seq结果的相关性。所有方法中的结果均表现出正相关,R2在0.53到0.88之间(图1F)。与miRXplore的结果一致,Lexogen 和 Norgen的相关性也是最差的。作者探讨了通过各方法特异的miRXplore结果来校正血浆样品中RNA-seq数据的偏倚。图1F为各方法内的RNA-seq结果的相关性,图1G为各方法间数据的相关性。结果表明,特异方法的结果偏倚在不同的样品间是存在的,在扣除已知的背景值后,能够使RNA-seq的值更加准确,不同方法间能够更好的进行比较。
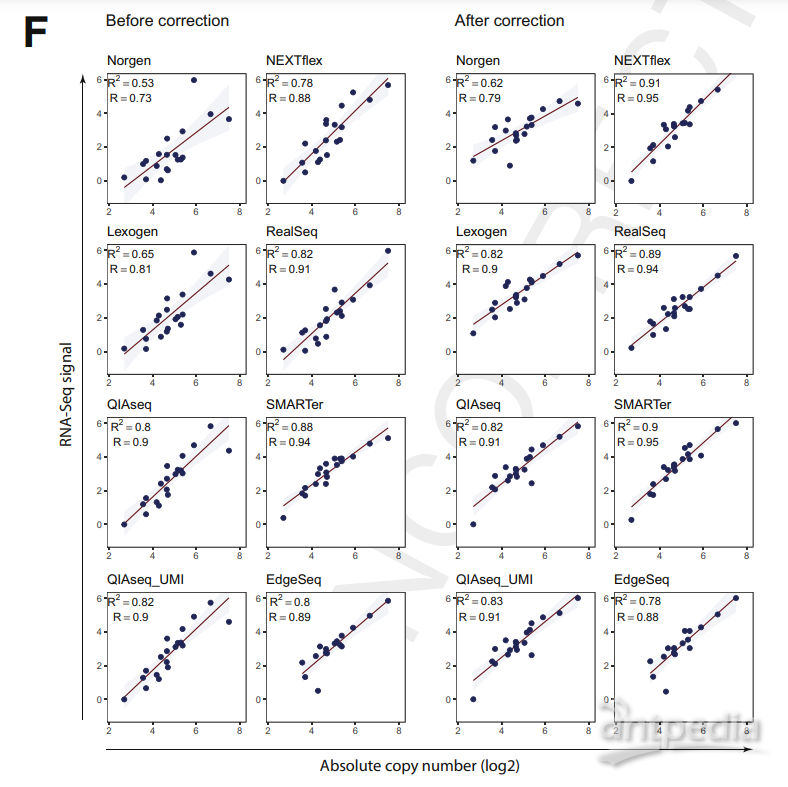
图1F: 血浆样品中small RNA-Seq 和RT-PCR (RT-qPCR) 定量结果基于miRXplore结果偏差校准前后的相关性

图1G:血浆样品各方法间在miRXplore进行校准前后的可重复性。P 值用双尾T检验。QIAseq UMI 代表deduplication后的数据, QIAseq 代表 non-deduplicated的数据
2.5 mapping统计

图2A: miRXplore 和 plasma样品的Mapping 统计
比较了各方法的Mapping 统计,miRXplore和血浆样品的结果有很大的不同。SMARTer的Mapping 率是明显较低的, EdgeSeq的Mapping 率最高,达到95%。在所有的方法中占比最大的miRNA-mapping reads都是很少的高丰度的miRNA。

图2B: 血浆样品中10个表达最高的miRNA。左边y轴表示血浆样品中raw reads 的比例,右边的y轴表示miRXplore 样品中技术偏差的差异水平。虚线表示预期值的两倍差异。
图2B列出了每个方法中检测到的表达量最高的10个miRNA,除了SMARTer 和NEXTflex,其余的方法中都有一个占比超过50%mapped reads的miRNA。虽然一些miRNA在所有的方法中检出都是最高的,如血红细胞特异的miR-451 和miR-16,与真实的量是一致的,但是其他的一些miRNA,如miR-10b 在 Norgen 和 Lexogen中由于结果偏倚导致了较高的差异(结果高出预期64倍以上)。
2.6 累积频率曲线分析

图2C: miRXplore 和血浆样品在线性和指数的累计频率。虚线分别表示50%和1%的累积频率。
为了评估在所有miRNA范围内的测序reads分布,进行了累积频率曲线分析(图2C)。数据表明EdgeSeq 和 SMARTer 结果好,而 Norgen 和 Lexogen 的表现最差。
2.7 测序深度和检出率分析

对不同测序深度和检测阈值的miRNA检出率进行分析(图2D)。大多数的方法在5M reads时检出接近饱和,而SMARTer 和RealSeq方法增加测序深度会获得更多的检出。EdgeSeq、QIAseq和NEXTflex方法检测到了最多数量的miRNA, Lexogen 和Norgen检测到的miRNA最少。EdgeSeq检测到的miRNA数量远超其他的方法。这不仅因为EdgeSeq有更高的mapping rate,而且也归因于较低特异性的杂交探针。

图2E: 小提琴图显示了在miRXplore 样品中的miRNA阳性和假阳性的水平
图2E显示了在miRXplore 样品中存在的miRNA(阳性)和不存在的miRNA(假阳性)的数据。在所有的方法中EdgeSeq方法显示有较高的假阳性率和较高的假信号强度,这也表明EdgeSeq方法在血浆样品中有较高的检出率部分原因可能是因为假阳性。

图2F: 各方法综合评估
结论
总结各个方法的结果,没有任何方法是好的。与其他的研究结论一致,作者的结果也表明无需接头连接的方法有最低的结果偏倚。EdgeSeq相较其他方法准确性更加显著,同时有更高的mapping 和检出率。EdgeSeq的另外的优点是其平台的自动化有更少的操作时间。EdgeSeq的缺点是有较高的分析成本和较低的特异性。
SMARTer是第二准确和最省力的方法,然而这种方法有最低的mapping率和最高的假reads和假isomiRs。
RealSeq的准确性与NEXTflex 和QIAseq类似。作者也发现环化的方法并不能消除结果偏倚。考虑到RealSeq是使用的两端接头连接步骤,结果也说明了连接是结果偏倚的显著来源。
在Lexogen、 Norgen 和 QIAseq 等3种传统的基于连接的方法中,Lexogen 和Norgen的结果并不是很好,与前人的结果一致。较强的连接偏倚导致miRNA的不平衡,较低的覆盖度和较低的检出率,因此需要更深的测序深度。QIAseq同样基于双端连接,但结果在大多指标中表现较好,作者认为这并不是因为QIAseq使用了UMI,而是因为该方法的细节是经过合理优化的。
除了方法间的比较,作者的数据也给出了改进在生物流体中进行small RNA 分析的几个内容:
首先,在血浆样品中miRNA分布有较高的不平衡性,较少的miRNA耗费了大多的reads,文库构建方法对miR-451 和 miR-16等含量较高的miRNA的检测是有利的。其次,合成的样品有已知背景的偏差,可以用于校准在真实生物流体样品中的准确性及不同方法间的相关性。最后,跟最近其他的研究结果相反,作者的结果表明如果UMI设计不合理,可能会导致miRNA数量产生严重的错误。
引用文献:
Peter Androvic, Sarka Benesova, Eva Rohlova, Mikael Kubista, and Lukas Valihrach: Small RNA-Sequencing for Analysis of Circulating miRNAs: Benchmark Study. The Journal of Molecular Diagnostics, VOLUME 24, ISSUE 4, P386-394, APRIL 01, 2022
DOI: https://doi.org/10.1016/j.jmoldx.2021.12.006


