
上海欧易生物医学科技有限公司
诚信认证:
工商注册信息已核实! 扫一扫即可访问手机版展台
扫一扫即可访问手机版展台
项目文章 | 欧易客户构建猴头菇高分辨率遗传图谱
- 研究背景 -
猴头菇(Hericium erinaceus)是一种著名的食用和药用蘑菇,可产生多种生物活性物质,例如多糖、萜类化合物、蛋白、凝集素和酚类化合物。在过去10来年中,猴头菇在生理和健康促进方面的特性被广泛报道,例如抗衰老、抗氧化、抗癌症等。
尽管其食用及药用的优点被广泛报道,聚焦于猴头菇的分子生物学以及遗传学特性的研究还很少。也因此,其遗传改良受限,进而阻碍了优良品种的育种。蘑菇育种的主要目的在于提高活性物的丰富度、产量、质量以及抗病能力,不过这些性状的改良通常面临着巨大的挑战,因为这些性状往往被数量性状位点(QTL)控制。
通过遗传图谱鉴定这些复杂性状相关的QTL,可以极大地促进标记辅助选择,加速育种进程,不过猴头菇目前尚无基因连锁图谱。遗传图谱绘制的一个重要条件就是遗传分离群体的发展。对于蘑菇来说,通过减数分裂得到的F1代例如单孢菌株(SSIs)是理想的遗传图谱绘制群体。而过去很少有在蘑菇的研究中用到了100个以上的减数分裂孢子进行研究,而这也限制了遗传图谱的准确性。
- 研究内容 -
猴头菇(Hericium erinaceus)因其引人注目的保健功效而引起广泛的关注。然而由于缺乏遗传和分子工具箱,猴头菇优良品质的选育受到了阻碍。本文作者对127个F1单孢菌株进行了重测序,构建了首(shou)个高分辨率的猴头菇遗传图谱。将重组bin作为标记物,得到了一个包含1174个bin的超高密度的遗传图谱(包含37082个SNP)。
新获得的遗传图谱总图距为1096.5 cM,平均每个bin为0.95 cM。通过利用bin标记作锚点,将scaffolds比对到这个遗传图谱上,遗传图谱和拼接的猴头菇基因组表现出高共线性。作者还将新获得的遗传图谱应用到数量性状位点(QTL)的定位上,获得了4个同单核体生长相关的QTLs。其中一个QTL,mgr1,占生长变异的12.1%,位于接合型A(MAT-A)位点附近。
总之,这一新构建的高分辨率遗传图谱可作为未来猴头菇遗传、基因组和育种研究的参考。
- 研究路线 -
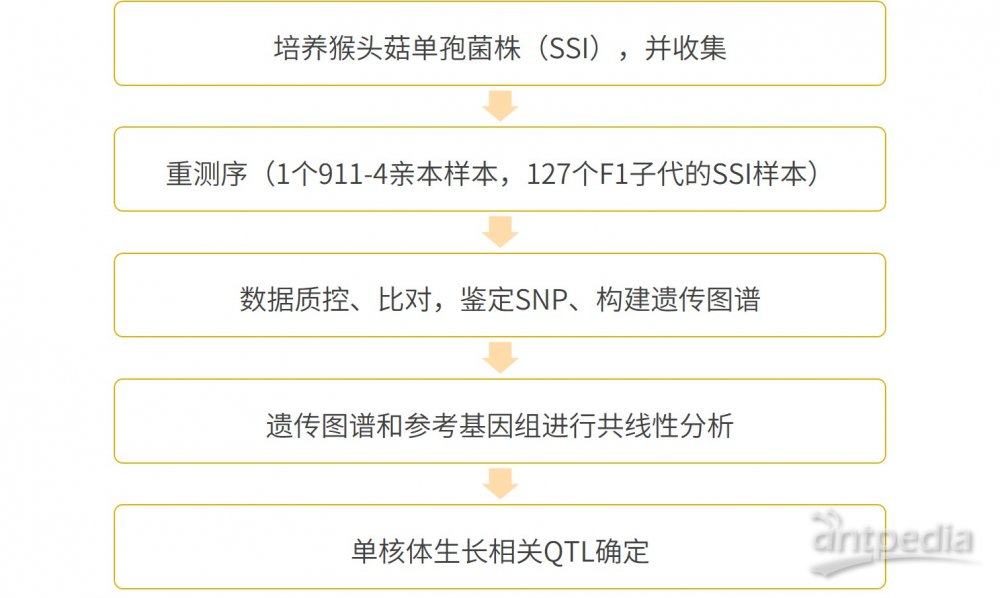
- 研究结果 -
1. 单孢分离群体的基因分型及遗传图谱的构建
作者用两个猴头菇单核体株CS-4和911-4通过接合获得了杂交系HeC9,收集HeC9担孢子并培养至萌发,而这些单核状态的SSIs作为图谱绘制的子代材料。为绘制遗传图谱,作者对911-4和127个SSIs进行了重测序。亲本单倍体911-4(覆盖度240×)共测到了10 G的clean reads,同时每个子代SSI(覆盖度最少为24×)也保证至少可以拿到1 G的clean data。在完成SNP过滤和bin校正后,作者一共获得了1522个bin,其中只有那些最小等位基因频率(MAFs)大于20%的bin被用于后续分析,最终得到了1174个bin(共包含37082个SNP),并构建了15个连锁群(LG)(见表1)。整个遗传图谱长度为1096.5 cM,覆盖了CS-4基因组上31.1 Mb。每个LG所含bin数量从15到117个不等,大小从18.6 cM到99.5 cM不等,平均间距小于1.0 cM,最大间隙在LG4处(7.86 cM)(见图1)。通过基因组注释和同源比对,作者确定了接合型基因的位置,MAT-A和MAT-B位于到LG1和LG9上(见图1)。
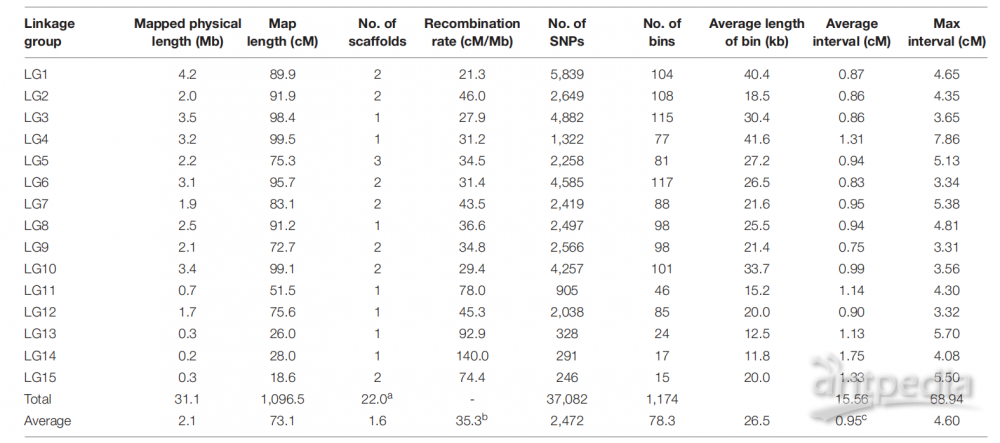
表1|猴头菇遗传图谱特点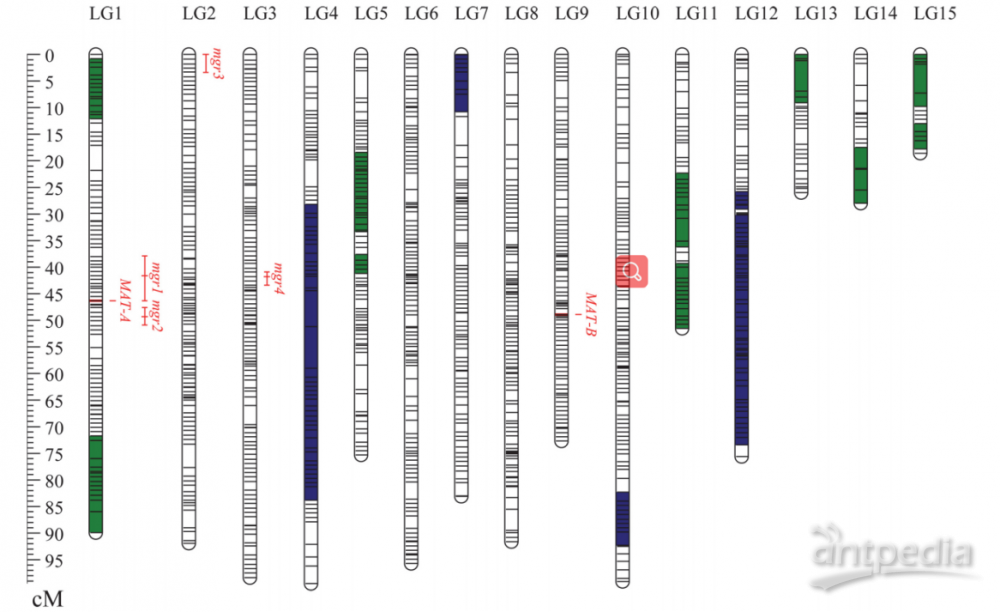
图1|猴头菇遗传连锁图上bin的分布
因为在人工绘制的双亲图谱群体中存在有限的交叉,对于一个给定的SSI,来自于同一单核体亲本的大基因组片段被校正出来(见图2)。在所有的SSIs,LG1中均为检测到两个以上的交叉发生,在几个SSI中,整个LG1片段遗传自CS-4且无交叉发生。相似的遗传模式也在其他LG中被检测到(见图2)。不过在所有15个LGs中,没有SSI仅继承自一方亲本。
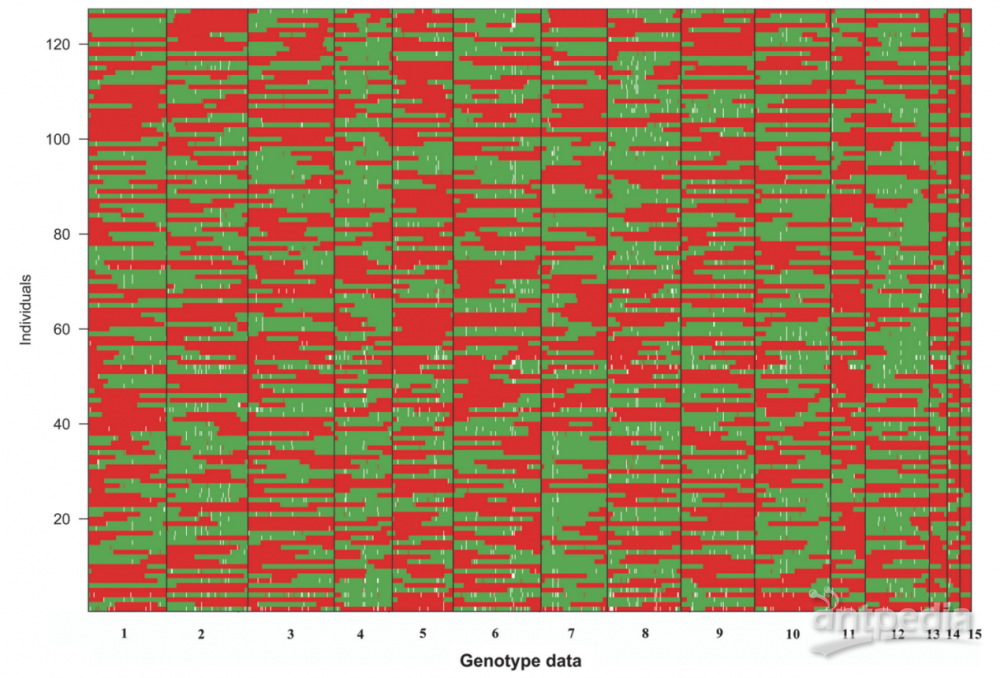
图2|SSIs基因型可视化
在SSI群体中,有242(20.7%)个bin被发现倾斜于一个亲本(p<0.001),其中110个bin倾斜于CS-4,其余的倾向于911-4。共220个有倾向性的bin被聚类为15个偏分离热点区域(SDRs),这些区域均包含3个及以上显示出偏分离的连锁标记物。这些SDRs分布于10个LG上,平均长度为15.4 cM(见表2)。
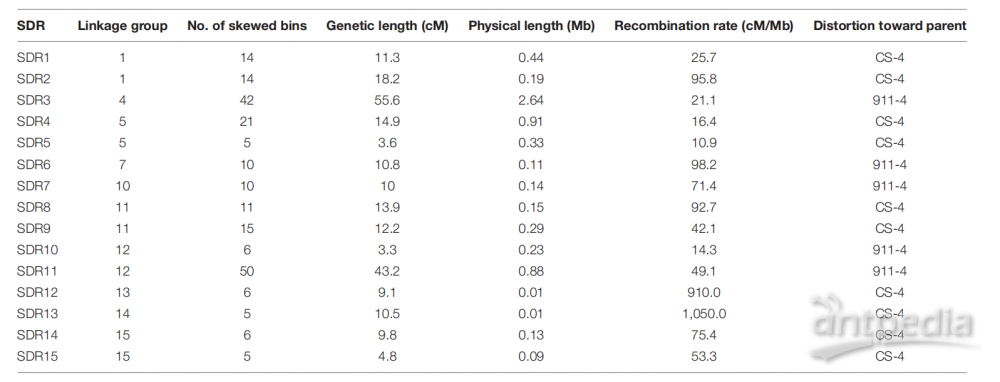
表2|猴头菇遗传图谱中检测到的偏分离热点区域
2. 基因组装配和遗传图谱比对
用bin标记物作为锚点,作者将22个scaffold(总长38.8 Mb)比对到遗传图谱上的15个LG。作者绘制了组装的scaffold和遗传图谱整合示意图(见图3),猴头菇的物理序列和遗传图谱呈现高度共线性关系。大多数maker在LG上的排序同基因组上的位置前后一致。除scaffold 3和6外,其余scaffold均比对到1个LG上。有7个scaffold和7个LGs正好对应(见表1)。
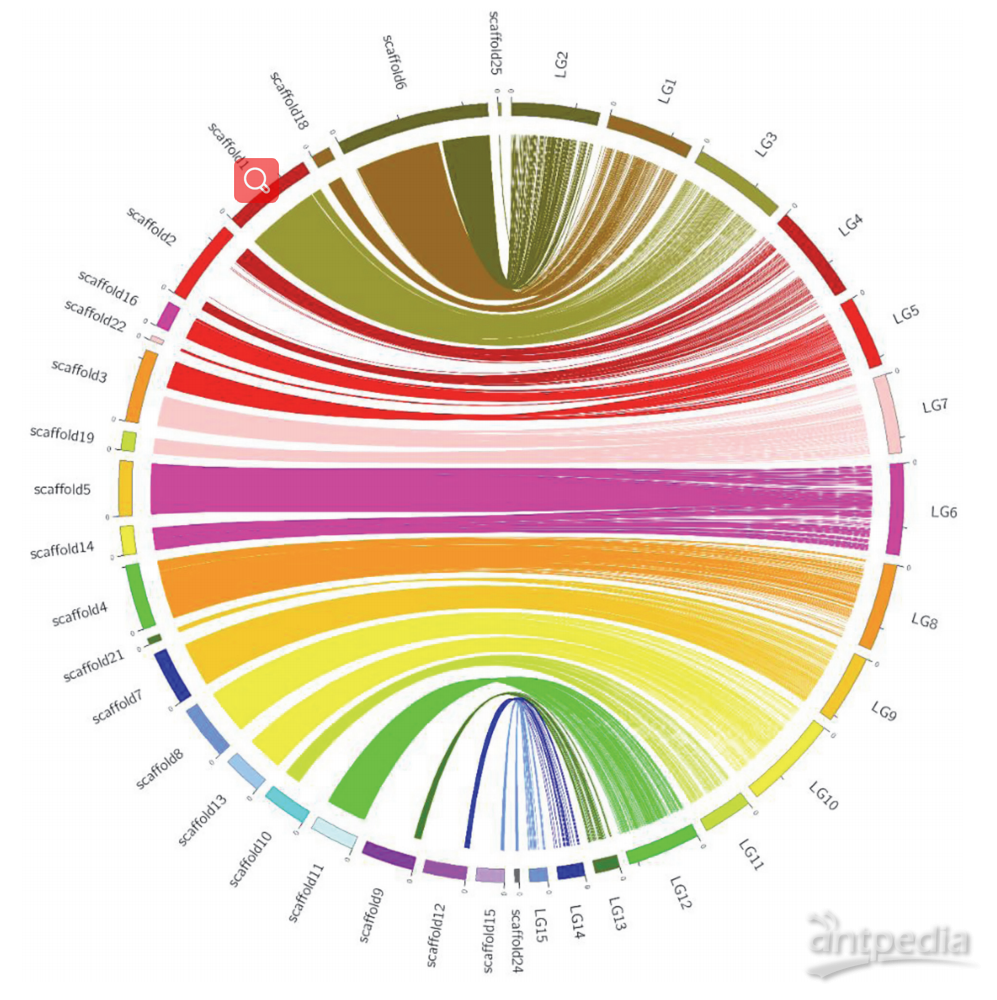
图3|猴头菇物物理图谱和遗传图谱的对应关系示意图
按总遗传图谱大小与被覆盖的物理长度之比计算,猴头菇的平均重组率为35.3 cM/Mb。各LG的重组率同物理长度呈现显著的负相关。为了计算染色体内重组率的变化,作者将检测到的SNP重新映射到这个遗传图谱中,然后将基因组分成不重叠的10kb窗口,计算重组率。最终作者共找到56个重组热点区域(大于平均重组率10倍以上的区域),且分布不均匀。一般重组率较高的区域是在LG端,这表明可能存在端粒区域。作者还统计了56个热点区域GC含量,发现热点区域GC含量明显较高。
3. 单核体增长的数量性状定位
CS-4和911-4两个亲本的单核体增长率(mgr)分别为3.2 和1.29 mm/天。而127个SSI的增长率范围是0.33-4.27 mm/天,平均为1.88 mm/天(见图4)。单方差ANOVA分析显示基因型对增长率有显著影响(p<0.01)。通过复核区间作图法(CIM),共有4个影响增长率的QTL,其中2个(mgr1 和mgr4 )同时被完备区间作图法(ICIM)证明。这4个QTL可解释39.2%单核体生长的变化,单QTL解释的表型变异范围为8.0%-12.1%(见表3)。这些QTL的物理置信区间长度从52 kb到2467 kb不等。分析这4个QTL置信区间的基因发现,mgr3和mgr4的小物理区间内,分别只包含22和19个编码基因。而在LG1上的mgr1和mgr2中,包含了100个基因 。一些被报道可能参与了真菌的营养生长和形态的基因(例如编码同源域转录因子、锌指转录调节因子和糖基转移酶的基因)也定位到这些QTL中。
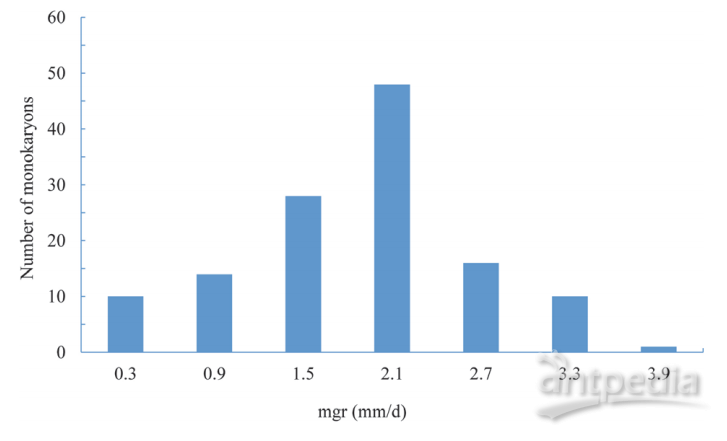
图4|SSI个体中单核体增长率分布
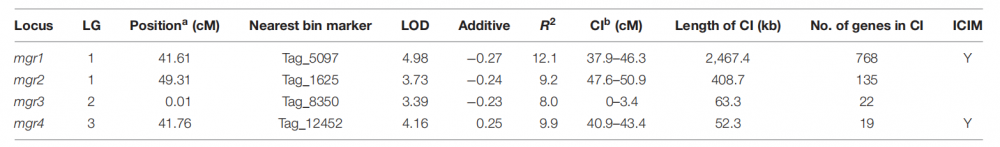
表3|猴头菇中控制单核体增长率的QTL
- 研究结论 -
本文利用高通量测序方法检测、鉴定了猴头菇中SNP的基因型,并绘制了高分辨率的猴头菇基因图谱。通过进行LG和scaffold的对比,发现猴头菇基因组与新完成的遗传图谱表现出高度的共线性。此外,本文还鉴定到了与猴头菇单核体生长高度相关的QTL。总之,这一新构建的高分辨率遗传图谱可作为未来遗传、基因组和育种研究的参考。
参考文献
Gong W, Xie C, Zhou Y, et al. A Resequencing-Based Ultradense Genetic Map of Hericium erinaceus for Anchoring Genome Sequences and Identifying Genetic Loci Associated With Monokaryon Growth. Front Microbiol 2020; 10:3129.
END
本文系欧易生物原创
转载请注明本文转自欧易生物


