诚信认证:
工商注册信息已核实! 扫一扫即可访问手机版展台
扫一扫即可访问手机版展台
编码和非编码RNA与蛋白结合鉴定
摘要
全转录组范围内鉴定能与蛋白结合的RNA(RBPs),是了解转录后基因调控网络的必要条件。然而RBPs蛋白组的研究主要局限于多聚腺苷酸的RNA与蛋白的结合,对于没有ploy-A尾的RNA(主要的是非编码RNA和RNA前体)几乎都没发现。本文介绍了一种点击化学(主旨是通过小单元的拼接,来快速可靠地完成形形色色分子的化学合成。它尤其强调开辟以碳-杂原子键(C-X-C)合成为基础的组合化学新方法,并借助这些反应(点击反应)来简单高效地获得分子多样性。)辅助的RNA互作捕获策略(CARIC),能够对RBPs进行无差别鉴定,不受RNA是否具有ploy-A尾的约束。CARIC主要是利用炔基尿苷类似物对RNA进行标记,并在活体内进行RNA-protein光照交联,然后与叠氮化物生物素进行点击化学反应,亲和富集后进行蛋白组分析。利用CARIC在人的宫颈癌细胞中鉴定到597个RBPs,包括130个之前未知的RBPs。这些新发现的RBPs可能是和非编码RNA结合的,因此发现了一些之前未知的非编码RNA参与的过程(例如蛋白酶体功能和中间代谢)。
材料方法
实验材料:人的宫颈癌细胞,胚肾细胞;
质粒构建和细胞转染:克隆宫颈癌细胞cDNA(hnRNPC,MBNL1,VDAC1,NME2),克隆质粒:VigoFect;几种已知RPBs用来验证CARIC技术成功率
CARIC分离出的RNA测序:Illumina HisEq 4000 PE150;
蛋白质谱检测:LC-MS/MS,Easy nLC 1000 system +Velos Pro Orbitrap Elite mass spectrometer;
质谱数据分析:MaxQuant version 1.5.5.1(原始数据分析),依靠人的蛋白数据(UniProt)Andromeda search engine进行蛋白查询;
CARIC RBPs验证:CLIP(交联免疫沉淀);
鉴定蛋白功能分析:等电点(ExPASy),密度分布曲线(MATLAB),蛋白结构域(Pfam),新陈代谢相关蛋白(Reactome)。
实验流程
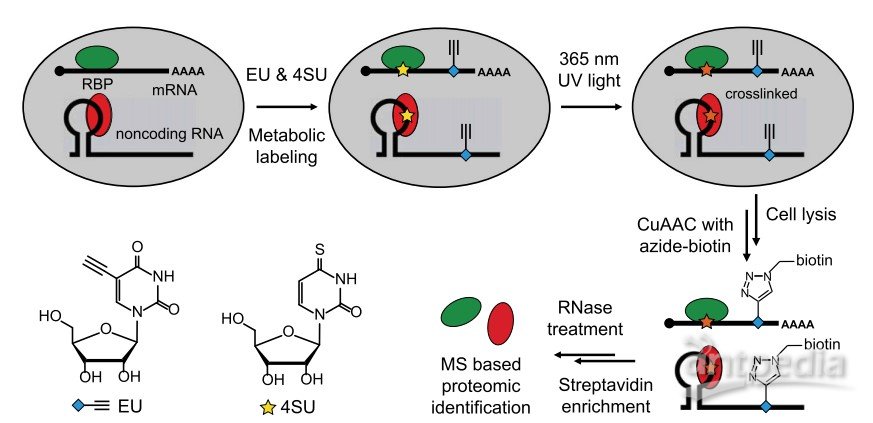
1、细胞内与蛋白结合的编码和非编码RNA(RPBs)首先用4SU(4-硫尿核苷)及EU(乙炔基尿苷)进行标记;2、365 nm UV光照射,使得4SU与RNA发生交联;3、细胞溶解后用铜催化的叠氮-炔烃环加成反应,再与标记到RNA上的EU发生交联;4、链霉亲和素磁珠富集标记了生物素的RPBs,用RNase处理这些RNPs(核蛋白颗粒),释放与RNA结合的蛋白,然后用LC-MS/MS对蛋白进行鉴定。
研究结果
1、CARIC实验的优化
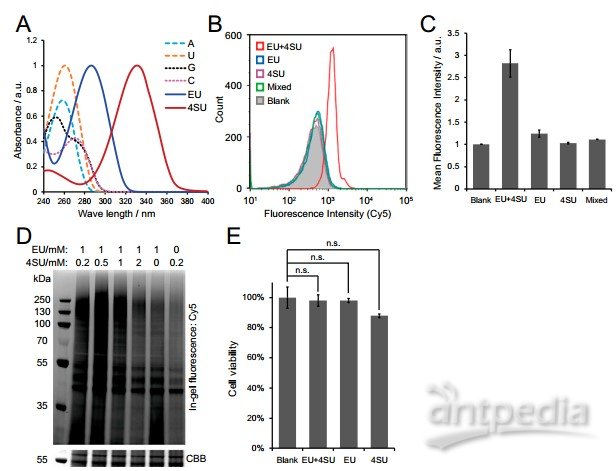
A.不同物质的紫外-可见光吸收光谱(365 nm紫外光只能激活4SU);B/C.流式细胞仪分析结果表明EU和4SU同时标记的细胞数量是最多的;D.对添加不同剂量EU和4SU的细胞进行紫外照射评估被RNP含量(1Mm EU+0.5 Mm 4SU处理下,>130kDa RNP含量最高);E.两种代谢标志物的添加对细胞的存活影响不明显。
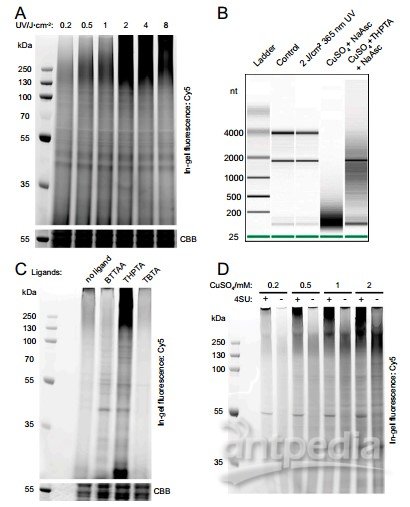
A.不同能量紫外光辐射下4SU和蛋白交联程度(2 J/cm2效果最好)B.紫外光照射对RNA降解的影响(2 J/cm2几乎不影响)C.几种不同催化剂配体评估对EU&4SU标记的效率(THPTA显著提高效率)D.0.5 Mm CuSO4标记效率最高;最优体系:1 mM EU+0.5 mM 4SU,2 J/cm2 UV,THPTA-CuAAC;
2、CARIC捕获结果的验证
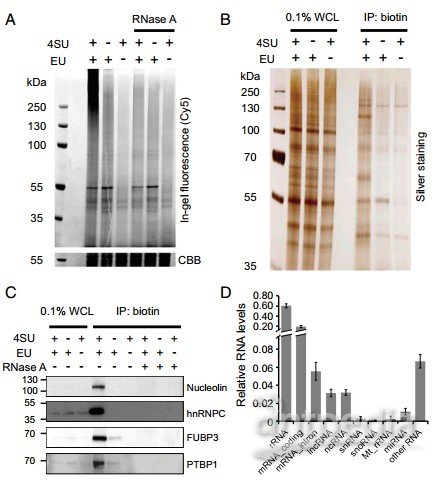
A. EU和4SU不同标记组合下RNPs含量(只有EU+4SU能检测到);B.链霉亲和素磁珠富集的标记后RPBs纯化产物,用RNase消化RNA之后,凝胶银染(WCL:全细胞溶解产物,IP:生物素免疫沉淀产物),EU和4SU双标条件下RPBs类型最丰富;C.WB检测CARIC捕获的几种蛋白(已知RPBs)的含量(表明CARIC能成功捕获RPBs);D.磁珠富集蛋白纯化后用蛋白酶K消化,然后进行RNA测序分析结合蛋白的RNA类型及水平(除了rRNA、mRNA以外非编码RNA也比较多)。
3.CARIC捕获RBPs的蛋白组鉴定(SILAC)
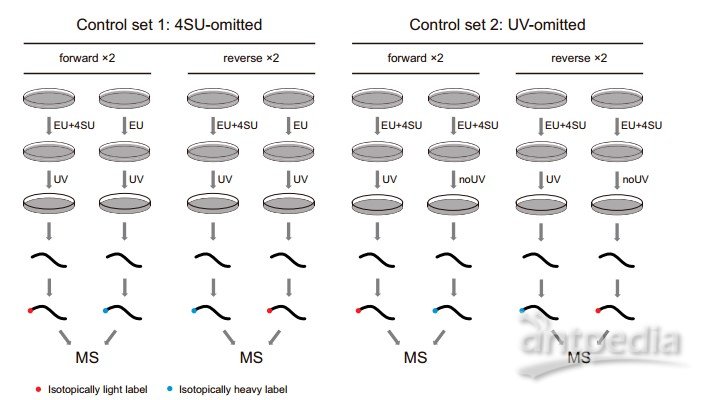
利用稳定同位素标记方法对分别缺失4SU和UV处理的对照组与正常处理(CARIC)进行蛋白质谱鉴定比较。
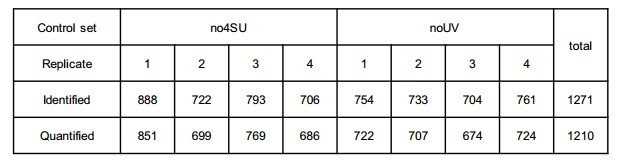
8个处理重复共定性到1271个蛋白,其中1210个被定量。
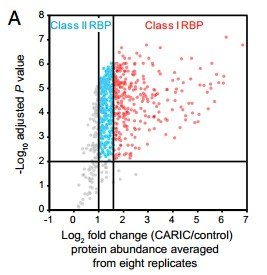
1210个被定量的蛋白中,691个蛋白(adjusted P < 0.01),正常处理和对照之间表达量差异在2倍以上的有597个RPBs,分为两类(ClassI>3=296,2)
4.CARIC RBPs与人类已知RBPs进行比较
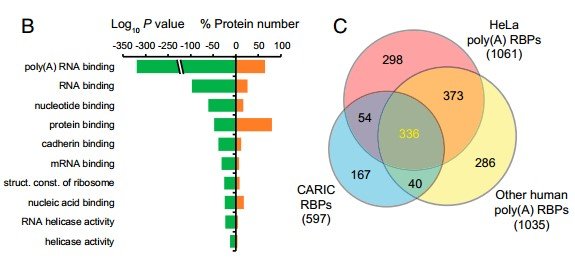
B. CARIC RBPs GO富集注释(富集最多的是protein binding,最显著的是poly(A)RNA binding);C. CARIC RBPs与人类已知poly(A)RPBs之间的重叠关系;
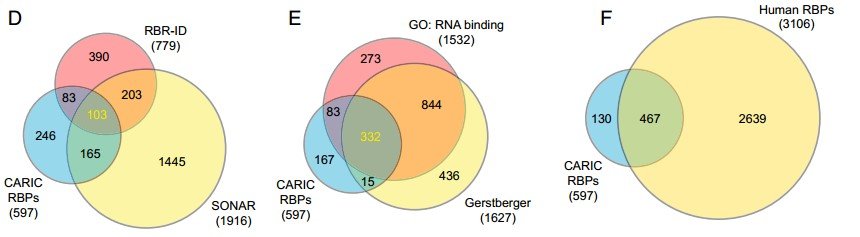
D. CARIC RBPs与另外两种鉴定RBPs方法的比较;E. CARIC RBPs与两种RBP数据库的比较;F. CARIC RBPs与人类已知RBPs的重叠关系(130个是新发现);
5.CARIC RBPs中RNA-Binding Activity蛋白的验证
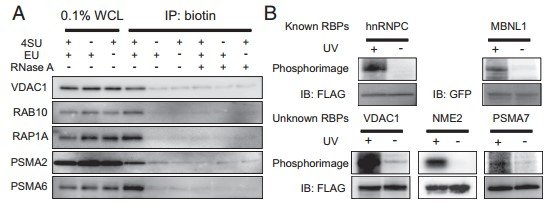
A.五种代表性的RNA-Binding Activity蛋白在全细胞溶解液和IP中不同标记及是否消化RNA条件下WB检测结果(CARIC能够捕获RBPs,丰度与两种标记有关,RNase A处理能降低富集丰度表明这些蛋白是和RNA互作或者交联);B.已知和未知RPBs用UV辐射交联FLAG或GFP,鉴定蛋白含量(已知RPB作为阳性对照)。
6.CARIC RBPs等电点,结合结构域,RNA特异性结合
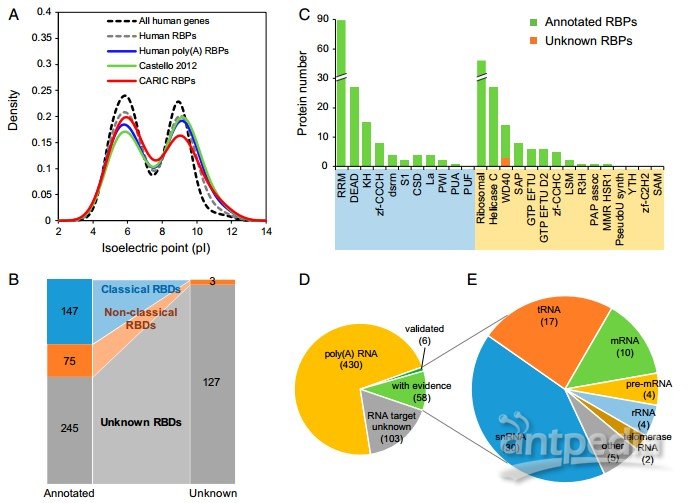
A.人类所有蛋白及不同来源RBPs等电点分布;B/C/D/E.功能已知和未知RBP数量及功能分类统计,结合RNA类型及数量统计(主要是poly(A)RNA);
7.CARIC 鉴定到的新RBPs解析
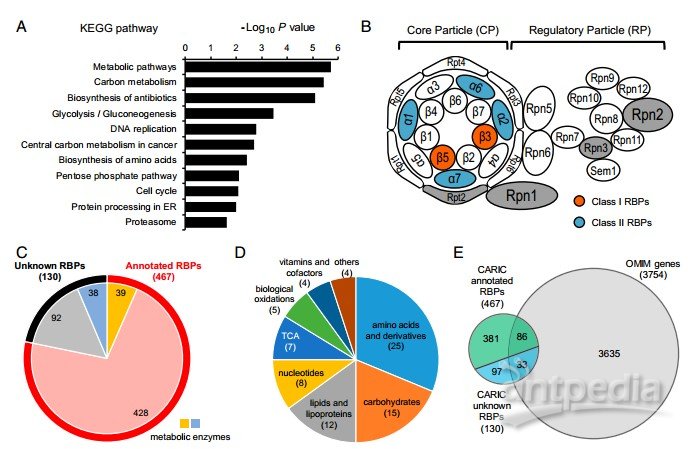
A.未知RBPs KEGG功能富集分析(其中一类比较富集的是“蛋白酶体”);B. RBPs中不同类型的蛋白酶体,橙色:class I RBP,蓝色:class II RBP,灰色:假定CARIC RBPs;C.已知和未知RBPs中代谢酶数量;D.特殊代谢通路中CARIC RBPs数量;E. CARIC鉴定到的RBPs与人类孟德尔遗传数据库中RBPs重叠关系(未知的重叠蛋白有33个)
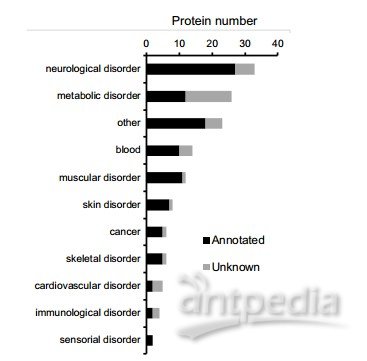
这些和OMIM重叠的未知蛋白与多种疾病都是相关的
研究亮点
1、传统RBPs蛋白组的研究主要局限于多聚腺苷酸的RNA,本研究利用点击化学辅助的RNA互作捕获策略(CARIC)克服了非编码RNA与蛋白结合鉴定的难题。
2、利用CARIC在人的宫颈癌细胞中鉴定到597个RBPs,与之前各种报道的RPB相比鉴定到130个之前未知的RBPs。并且利用WB,CLIP实验验证了部分未知RBP的准确性。针对鉴定到的RBP,分别利用SILAC和转录组测序,描述了蛋白的和RNA定性定量。
参考文献
Transcriptome-wide discovery of coding and noncoding RNA-binding proteins[J].2018.PNAS