
诚信认证:
工商注册信息已核实! 扫一扫即可访问手机版展台
扫一扫即可访问手机版展台
文献解读 | 大片段建库方法创新结合全基因组测序,临床应用前景广阔
2019年6月7日,香港中文大学和华大团队在DNA RESEARCH【1】上在线发表Development of couplingcontrolled polymerizations by adapter-ligation in mate-pair sequencing fordetection of various genomic variants in one single assay一文。该研究提供了一种成本合理且高效率的大片段文库构建方法,该方法可用于人类疾病中各种基因组变异的检测,具有广阔可期的临床应用前景。作者认为在单分子测序技术特别成熟之前,高通量测序技术大片段建库方法仍有巨大的市场,但是需要更为简化的流程。
我们先听听文章的作者们是怎么评论这一研究成果的:
研究摘要
该研究主要基于3’ branch ligation高效的连接反应和两种可控的酶聚合反应
作者“声音”
该研究展示了在提高建库效率和降低成本的情况下,大片段建库结合全基因组测序,可作为一种检测多类型疾病变异的整体解决方案。
—香港中文大学妇产科学系,董梓瑞博士
CP-AL这种全新mate pair大片段建库方式提高了全流程效率,降低成本和建库时间,简易的操作性能真正实现全流程的自动化,同时也可有效避免小片段污染问题和提高mate-pair reads比率。大片段建库和全基因组测序结合能检测SNVs,CNVs,SVs等多类遗传变异,具有广阔的临床应用前景。此技术已申请PCT专利。
—华大智造应用研发中心-新技术研发
下面,让我们一起了解这篇文章的具体内容:
(控制切口平移和引物延伸),结合在接头序列上形成的14nt的反向互补序列双链成环技术,开发出一种无需切胶、无需生物素捕获的大片段Mate-pair文库构建方法。
通过后期测试和验证,此方法与传统的平末端环化方法相比,可以显著提高DNA环化效率(4倍),可以几乎完全去除DNA短片段的污染(39.3倍)。最终在同样测序深度的情况下,增加50%的物理覆盖率,同时基因组/外显子覆盖度较为均一。
由于测序模板的长度可以通过调整dNTP的量来精确控制,该研究比较了不同测序长度对染色体异常样本中基因组变异的检出情况,证明更长的测序长度将进一步提高基因组变异的检出率和诊断率。
最后,该研究通过对大片段文库进行全基因组测序(30X),同时完成单核苷酸突变、拷贝数变异和染色体结构变异的检测,初步证明其作为一种检测多类型疾病变异的整体解决方案,促进了对人类疾病的遗传诊断。
研究背景
人类基因组变异包括single-nucleotide variants (SNVs), copy-number variants (CNVs) and structural variants (SVs)。 人类疾病中SNVs和CNVs是公认的致病因素,同时越多的研究也证明了检测SVs的重要性。
儿科遗传病学诊断常用的方法是WGS(小片段文库),至少有8.1%的异常在WGS检测中存在漏检。其中一个原因是CNVs和SVs存在大量重复区域,不适合用小片段WGS检测。因此开发了多种大片段Mate-pair方法用于检测SVs。但这些方法均需要生物素标记或切胶,存在数据浪费、假阳性高、操作繁琐等缺陷,限制了它们在临床上的应用(表1)。最后单分子测序虽然读长长,但是高成本和测序错误率高阻碍了其应用,因此团队开发了一种可控的酶聚合反应,无切胶和无生物素捕获的大片段Mate-pair文库,解决上述问题。
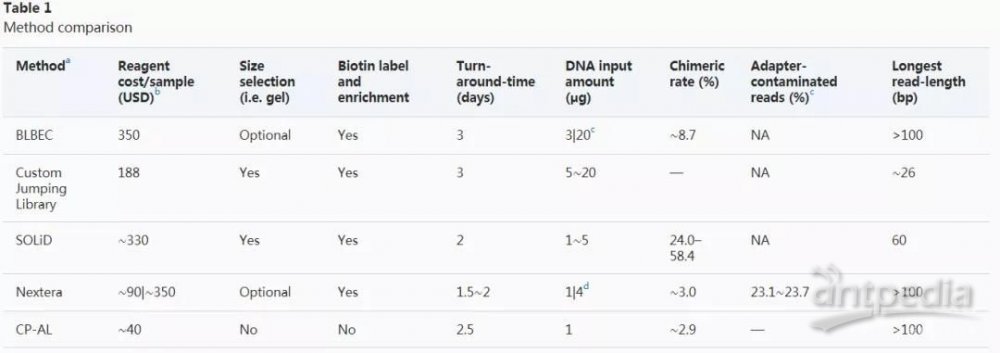
表一:大片段方法比较
研究方法

1ug gDNA打断后片段3-8Kb 大小,打断后片段加入接头Ad1, PCR(带U)、USER酶切,粘性末端双链环化(1bp切口),通过限制性切口平移(CNT)控制切口的长度,再加入Ad2_3’接头,通过限制性引物延伸(CPE)控制模板延伸的长度,再加入Ad2_5’接头,最后PCR形成两端带Ad2接头序列、中间为Ad1接头序列的mate-pair文库。
技术核心:
1.片段长度控制:通过控制反应条件、试剂用量等来实现对切口平移和引物延伸长度的控制;
2.特殊的接头结合方式:Branch Ligation可以使接头与双链中的一条链结合。

研究结果
研究结果表明:
临床样本验证CP-AL方法可行

图A-F:6个样本测序结果与ref GC偏差小,一致性高(r 在人参考基因组计算的每个窗口中的GC百分比与比对读数的GC百分比之间> 0.98)、
图G:CP-AL方法小于<1kb 的片段只有~0.7%,而生物素标记方法为27.5%和Nextera方法为24.4%(小片段比例小,污染少)
图H:与CP-AL方法相比,使用常规大片段建库方法观察到物理覆盖率降低约33%(物理覆盖完整)
CP-AL方法不同测序读长检测SVs效果
PE100测序:断点位置的精确检测对遗传诊断的检测非常重要
PE100精确检测到5个易位中26个重排(100%);PE50诊断率为92.3%(24/26);PE26诊断率为84.6%(22/26)
从诊断率来看,读长越长可以更全面地检测结构重排
六个样本中,CP-AL方法和CMA方法检测的CNV具有高度一致性

染色体结构重排
A:不同颜色的线代表不同样本结构重排情况。红箭头代表PE26,蓝箭头代表PE50
B:样本3染色体结构重排的情况,箭头表示基因的方向(PE100)
C:样本5染色体结构重排的情况,箭头表示基因的方向(PE100)
CP-AL方法:可以同时检测SNVs,CNVs和SVs
Trisomy2样本证明了CP-AL方法CNVs检测结果的一致性
Trisomy2样本SVs的分析同时可以提供重复和缺少的检测分析
CP-AL方法可以做所有变异检测的整合,这是标准WGS暂时无法做到

Trisomy2样本全基因组变异分析
从外到内:最外圈表示24条染色体显带,第二圈表示区域覆盖深度和copy ratio比率,第三圈和第四圈染色体copy ratio比率情况,其中2号染色体全长为蓝色代表拷贝数加倍;最内圈红线代表染色体区域缺失,蓝色代表染色体区域重复;黑线表示:SVs分析中染色体6和X存在易位。
讨论
本文提供了一种成本合理且高效率的大片段文库构建方法,它提高了reads利用率,使基因组/外显子组覆盖更均一,并且成本是当前主流建库方法成本的一小部分。该方法可用于人类疾病中各种基因组变异的检测。并且作者认为在单分子测序技术特别成熟之前,高通量测序技术大片段建库方法仍有巨大的市场,但是需要更加简化的流程。
本文对以下方面进行讨论:
首先,CP-AL具有DNA投入量少;无需切胶纯化,可降低人工成本;无需生物素标记磁珠标记捕获,降低整体成本和短序列 (inward sequences) 残余比例;无需第二次打断;基因组/外显子组覆盖均一;chimeric rate低;CP-AL方法可替代标准WGS检测八项优点。
但基因变异的临床实际意义,仍需要进行全面的临床评价。此外,尽管该方法成本低/操作简单使用于常规实践,但仍存在改善的空间: 包括引物包含dUTP,会降低>10kb的长片段扩增效率,可能会限制SVs的检测;读长需要增长,为了检测精度更准确;酶切打断替换物理打断。
数据来源
本次研究中所有全基因组测序原始数据均保存在国家基因库生命大数据平台(CNGBdb),项目编号:CNP0000078。
国家基因库生命大数据平台(China National GeneBank DataBase, CNGBdb)是一个为科研社区提供生物大数据共享和应用服务的统一平台,基于大数据和云计算技术,提供数据归档、知识搜索、计算分析、管理授权等数据服务。
参考文献
Dong Z, Zhao X, Li Q, Yang Z, Xi Y, Alexeev A, et al. Development ofcoupling controlled polymerizations by adapter-ligation in mate-pair sequencingfor detection of various genomic variants in one single assay. DNA Res. 2019.Epub 2019/06/08. doi: 10.1093/dnares/dsz011. PubMed PMID: 31173071.
